Inhaltsverzeichnis:
- Schritt 1: Verwenden von Brain Box
- Schritt 2: Intro: Querschnitte analysieren
- Schritt 3: Einrichten einer Funktion: LevelCurveTracings.m
- Schritt 4: Entwicklung des Bound-Algorithmus: Bounds finden
- Schritt 5: Entwicklung des gebundenen Algorithmus: Generieren eines äußeren gebundenen Arrays
- Schritt 6: Entwicklung des gebundenen Algorithmus: Arbeiten mit Mittelpunkt
- Schritt 7: Entwicklung des gebundenen Algorithmus: Wenn ein Schwerpunkt nicht zentriert ist
- Schritt 8: Entwicklung des gebundenen Algorithmus: Interferenz von Löchern
- Schritt 9: Entwicklung des Bound-Algorithmus: Lokalisieren von Löchern, Finalisieren von Brain Bounds und Hole Bounds
- Schritt 10: Daten protokollieren: Funktion PatientFiles.m
- Schritt 11: Daten in Dateien protokollieren
- Schritt 12: Protokollieren von Daten: Anzeigen eines Diagramms des Gehirnvolumens über die Zeit
- Schritt 13: Lücken in Subplots schließen: Subplotclose.m
- Schritt 14: Der Hauptcode: Alles löschen und Eingabeaufforderungen
- Schritt 15: Der Hauptcode: Stapelverarbeitung der Bilder
- Schritt 16: Der Hauptcode: Padding
- Schritt 17: Der Hauptcode: Grenzen bestimmen
- Schritt 18: Der Hauptcode: Bestimmen des richtigen Z-Maßes
- Schritt 19: Der Hauptcode: Bestimmung der X- und Y-Koordinaten
- Schritt 20: Der Hauptcode: Zeichnen einer dreidimensionalen Struktur, Finden des Volumens und Protokollieren von Daten
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-31 10:15.
- Zuletzt bearbeitet 2025-01-23 12:52.
Der Vorstoß in die Grenzen eines längeren menschlichen Lebens hat den Anstieg von Krankheiten hervorgebracht, die Zivilisationen vor unserer nicht gesehen haben. Unter diesen waren 2017 etwa 5,3 Millionen lebende ältere Amerikaner von Alzheimer betroffen, oder etwa 1 von 10 älteren Amerikanern (https://www.alz.org/facts/) und unzählige andere mit Demenz. Um den Kampf zu unterstützen, zu verstehen, was unsere Älteren belastet, wird dieser Code zukünftige Forscher und neugierige Forscher mit der Fähigkeit ausstatten, das Gehirnvolumen im Laufe der Zeit zu verfolgen.
Schritt 1: Verwenden von Brain Box
Um die Brain Box zu nutzen, braucht man nur Folgendes:
- MRT-Scans eines Gehirns und Name und Format solcher Dateien (sollten alle ungefähr die gleichen Abmessungen haben)
- Länge eines Scans
- Abstand zwischen den einzelnen Schichten (MRT-Scan)
- Patientenname (Bei der Eingabe keine Leerzeichen einfügen und Vor- und Nachname bitte groß schreiben, z. B.: VornameNachname)
Und daraus hat man die Fähigkeit, die Entwicklung des Gehirnvolumens einer Person im Laufe der Zeit zu verfolgen. Somit können Zahlen zu Alzheimer-Trends mit dieser Software verfolgt werden. Die Länge, die wir im Versuch verwendeten, betrug 180 mm für die Länge eines Scans und 5 mm für den Abstand zwischen den MRT-Scans, basierend auf Durchschnittswerten.
Die Anwendung der Brain Box muss jedoch nicht auf diese eine Aufgabe beschränkt sein. Wenn die Querschnitte eines bestimmten Festkörpers fotografiert werden, wie ein Tumor an sich, können auch die Trends der Volumenänderungen für diese in der Software verfolgt werden.
Schritt 2: Intro: Querschnitte analysieren
In dreidimensionalen Strukturen werden die zweidimensionalen Ebenen, aus denen diese bestehen, Querschnitte genannt. Stellen Sie sich vor, dass ein Stapel Papier ein rechteckiges Prisma bildet, dann wäre jedes Stück Papier ein Querschnitt des Papiers. Bei der Vorstellung des Gehirns wenden wir den gleichen Gedankengang an. Die MRT (Magnetresonanztomographie) (siehe Informationen zur MRT) erfasst die Querschnitte des Gehirns, und aus der Verwendung der Grenzen, die in jeder bereitgestellten "Schicht" des Gehirns definiert sind, können wir eine Struktur konstruieren, um das Volumen des Gehirns zu modellieren und zu bestimmen. Wir müssen jedoch zuerst eine Funktion erstellen, die Informationen über solche Grenzen liefert.
Schritt 3: Einrichten einer Funktion: LevelCurveTracings.m
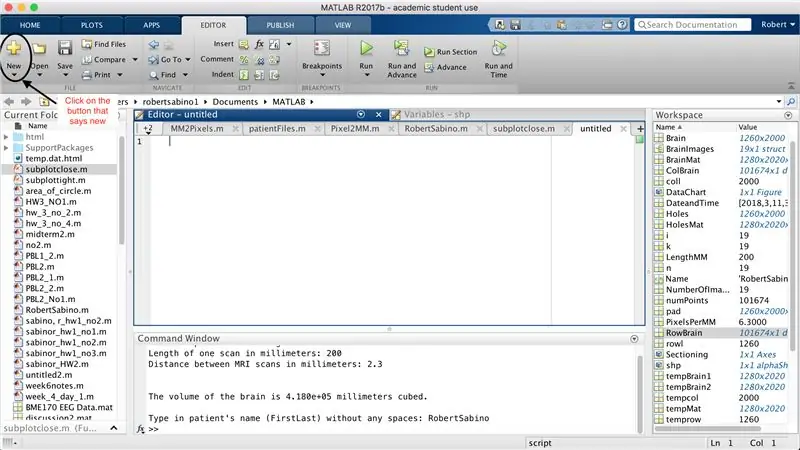
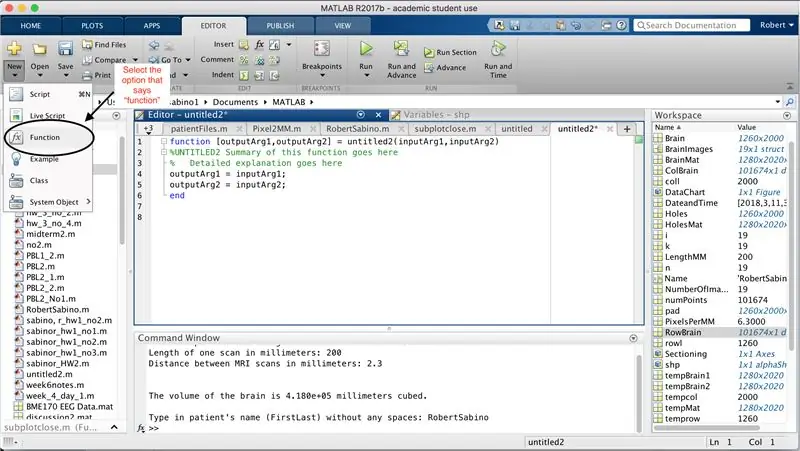
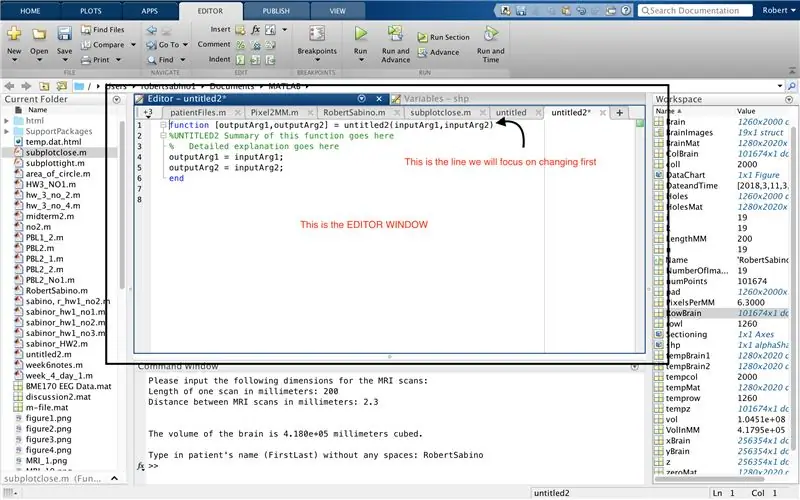

Stellen Sie zunächst sicher, dass auf Ihrem Computer MATLAB_R2017b heruntergeladen wurde (hier herunterladen) und öffnen Sie MATLAB. Klicken Sie in der MATLAB-Oberfläche auf die Schaltfläche in der oberen linken Ecke des Fensters mit der Aufschrift "Neu" mit einem fetten gelben Pluszeichen und wählen Sie die Option "Funktion", um im Editorfenster ein Leerzeichen zu öffnen, das dem im drittes Bild. Wir werden uns darauf konzentrieren, die erste Zeile zu ändern, um die Funktion einzurichten. Wo "outputArg1" steht, ersetzen Sie es durch "brain", "outputArg2", um "Löcher" zu sagen, "untitled2" zu "exp2" und "inputArg1" zu "image" und löschen "inputArg2". Sie haben jetzt eine Funktion, die mit "exp2" aufgerufen wird, ein Argument "image" nimmt und die Grenzen von "brain" und "holes" ausgibt. Die erste Zeile der Funktion sollte der im vierten Bild dargestellten Zeile ähneln. Löschen Sie den gesamten Code unter dieser ersten Zeile.
Schritt 4: Entwicklung des Bound-Algorithmus: Bounds finden

Geben Sie den Code wie folgt unter der Zeile ein. Dieser Abschnitt der Funktion führt zeilenweise die folgenden Schritte aus.
- Laden Sie das Bild "image" in die Variable "mri".
- Verwandeln Sie "mri" in ein Bild, das aus Werten in einem Bereich von Zahlen bis Einsen und Nullen (auch Binarisierung genannt) basierend auf einem festgelegten Schwellenwert besteht. Wenn der Wert in einem Pixel gleich oder größer als 0,1 ist, wird er auf eins gesetzt, wenn nicht, wird der Wert an diesem Pixel auf null gesetzt.
- Die folgenden vier Zeilen verwandeln 10 Spalten und Zeilen an den Rändern der MRT-Scanschicht in Nullen, um zu vermeiden, dass falsche Werte als Begrenzungen gelesen werden (wie aus dem Experimentieren mit dem Code gelernt).
- In der letzten Zeile verfolgt bwboundaries die Grenzen des binarisierten Bildes "mri" und setzt es gleich "b", ein Array mit den Elementen, deren Indizes denen der auf eins gesetzten Grenzen entsprechen.
Schritt 5: Entwicklung des gebundenen Algorithmus: Generieren eines äußeren gebundenen Arrays
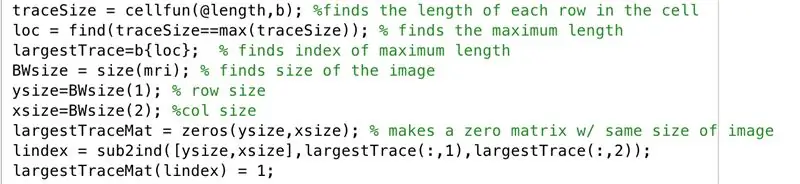
Folgen Sie im Editorfenster dem folgenden Code im Bild. In diesem Abschnitt des Codes wird die folgende Zeile für Zeile ausgeführt.
- Finden Sie die Länge jeder der Zeilen des binarisierten Bildes "b" (cellfun wendet die Funktionslänge auf jede Zeile an).
- Setzen Sie "loc", um die maximalen Längen zu speichern.
- Finden Sie den Index der maximalen Länge, der in "largestTrace" gespeichert werden soll.
- Finden Sie die Größe des Bildes "mri", das aus der gleichen Größe wie "b" besteht, und stellen Sie "BWsize" ein.
- Ermitteln Sie die Anzahl der Zeilen im Array des Bildes, setzen Sie sie auf "ysize".
- Finden Sie die Anzahl der Spalten im Array des Bildes, eingestellt auf "xsize".
- Erzeuge das Array "largestTraceMat", eine "ysize" mal "xsize" Matrix von Nullen.
- Finden Sie den äquivalenten Index aus den tiefgestellten Werten, die dem entsprechen, wo die größten Trace x-Werte und y-Werte waren, speichern Sie im Vektor "lindex".
- In der Nullmatrix "largestTraceMat" verwandeln Sie die Elemente an den Indizes, die Indexwerten entsprechen, die als Elemente in "lindex" gespeichert sind, in Einsen.
Somit hat das logische Array "largestTraceMat" den größten begrenzten Bereich des gegebenen Gehirnscanquerschnitts, der als Einsen mit einem Hintergrund von Nullen aufgetragen wird
Schritt 6: Entwicklung des gebundenen Algorithmus: Arbeiten mit Mittelpunkt

Als nächstes müssen wir testen, ob der Querschnitt aus mehr als einer Region (der größten) besteht. Durch Testen der Ausrichtung des Schwerpunkts der größten Region können wir sehen, ob es eine zusammenhängende Region gibt, die einen stärker zentrierten Schwerpunkt ergeben würde, oder ob mehrere Regionen möglich sind.
- Verwenden Sie "regionProps", um Informationen zu den vorhandenen Zentroiden zu finden, setzen Sie sie gleich dem Strukturarray "tempStruct"
- Bilden Sie Array "centroids" mit Daten aus dem Feld "centroid" vertikal verkettet
- Nehmen Sie die Werte der zweiten Spalte von "centroids" (die horizontalen Dimensionskoordinaten)
- Führen Sie einen Filter aus, um die Ausrichtung des Schwerpunkts zum horizontalen Mittelpunkt zu überprüfen
Schritt 7: Entwicklung des gebundenen Algorithmus: Wenn ein Schwerpunkt nicht zentriert ist
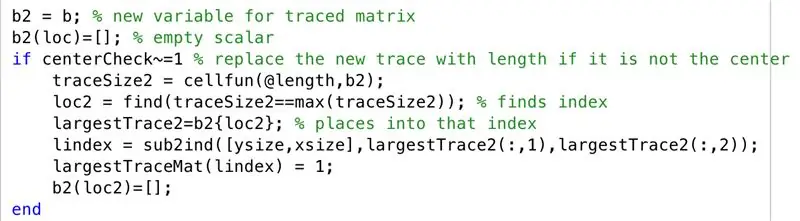
In dem Szenario, in dem der Schwerpunkt des größten Spurbereichs nicht zentriert ist, gehen wir die folgenden Schritte durch. Wie wir bei den MRT-Scans beobachtet hatten, bestand die Tendenz, dass im Querschnitt nicht aneinandergrenzende Hemisphären des Gehirns dargestellt wurden.
- Setzen Sie die verfolgte Matrix auf eine neue Variable "b2"
- Initialisieren Sie die leere Matrix "b2", mit einem durch "loc" indizierten Satz
- Erstellen Sie eine Bedingung, wenn ein Schwerpunkt nicht zentriert ist (d. h. ein Layer mit mehreren Regionen)
- Legen Sie eine neue Spurgröße fest, die für jede Zeile gefunden werden soll (traceSize2)
- Setzen Sie "loc2", um die Indizes zu finden, bei denen Grenzen vorhanden sind
- Lassen Sie Zellen, die durch "loc2" in "b2" angegeben sind, gleich "largestTrace2"
- Konvertieren Sie die Indizes in Indizes, setzen Sie auf "lindex"
- Ändere Elemente, die "lindex" in "largestTraceMat" entsprechen, auf 1
- Initialisieren Sie die leere Matrix "b2", mit einem durch "loc2" indizierten Satz
Schritt 8: Entwicklung des gebundenen Algorithmus: Interferenz von Löchern

Bei der Behandlung von Löchern verfolgten die in "b2" gespeicherten Werte andere Strukturen als die größte Spur, und wenn diese auf ein ausgefülltes Formular von "largestTraceMat" aufgetragen werden, werden die Löcher in den Gehirnregionen sichtbar.
- Erstellen Sie das Array "filledMat", das eine ausgefüllte Form von "largestTraceMat" ist.
- Erstellen Sie das Array "interferenceMat", ein "ysize" by "xsize" Array von Nullen
- Array "interferenceloc" erstellen, um die Werte von "b2" zu speichern, vertikal verkettet
- Erstellen Sie das Array "lindex", um die Indizes zu speichern, die "interferenceloc" entsprechen
- Für Indizes in "interferenceMat", die "lindex" entsprechen, setzen Sie den Wert auf 1, wodurch ein anderer begrenzter Bereich entsteht
Schritt 9: Entwicklung des Bound-Algorithmus: Lokalisieren von Löchern, Finalisieren von Brain Bounds und Hole Bounds

- Array "tempMat" gleich "interferenceMat" plus "filledMat" setzen und so jeden Wert in der Matrix zueinander addieren
- Setzen Sie das Array "holesLoc" gleich den Indizes, bei denen "interferenceMat" und "filledMat" beide gleich eins waren
- "holesMat" als Nullmatrix der Dimensionen "ysize" x "xsize" einrichten
- Setzen Sie Indizes in "holesMat", die gleich "holesLoc" sind, als Einsen
- Setze "Gehirn" auf "größteTraceMat"
- Setze "holes" auf "holesMat"
Mit der Feststellung, dass die Werte der hinzugefügten Matrizen gleich 2 waren, wurden die Lochpositionen leicht gesichert und auf eine leere Matrix aufgetragen.
Schritt 10: Daten protokollieren: Funktion PatientFiles.m

Ähnlich wie bei der Einrichtung der letzten Funktion, klicken Sie auf die Schaltfläche in der oberen linken Ecke des Fensters mit der Aufschrift "Neu" mit einem fetten gelben Pluszeichen und wählen Sie die Option "Funktion", um im Editorfenster ein Leerzeichen zu öffnen, das ähnelt dem im dritten Bild. Löschen Sie in der ersten Zeile die Ausgabematrix und ersetzen Sie sie lediglich durch "output", ersetzen Sie "untitled2" durch "patientFiles", löschen Sie alle Eingabeargumente und folgen Sie stattdessen der im vierten Bild der Codezeile angegebenen Formatierung. Die erste Zeile dieser Funktion sollte der Formatierung des Bildes entsprechen.
Schritt 11: Daten in Dateien protokollieren
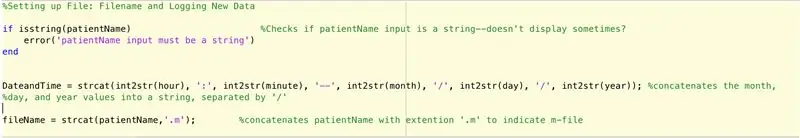

Um eine Datei einzurichten, um die von der Hauptfunktion gefundenen Daten (noch zu beschreiben) zu protokollieren, müssen wir diese Schritte ausführen (wie durch den Code zeilenweise vorgeschrieben).
- Prüfen Sie, ob die Eingabe für patientName eine Zeichenfolge ist.
- Wenn es sich nicht um eine Zeichenfolge handelt, zeigen Sie an, dass die Eingabe von patientName eine Zeichenfolge sein sollte.
- Beenden Sie die if-Anweisung (Fehler vermeiden).
- Richten Sie eine String-Anweisung "DateandTime" ein, die das folgende Format hat: Stunde:Minuten--Monat/Tag/Jahr.
- Setzen Sie die Variable fileName auf Folgendes: patientName.m.
Nun zum nächsten Abschnitt der Funktion: Existiert bereits eine Datei mit diesem Namen?
1) Angenommen, die Datei mit diesem Namen existiert bereits:
- Führen Sie die Datei aus, um die Werte aus der Vergangenheit in die Warteschlange zu stellen
- Fügen Sie die "DateandTime"-Daten der aktuellen Iteration als neue Zelle in das Zellen-Array von x-Werten (Index-Ende+1) ein
- Fügen Sie den aktuellen "brainVolume"-Wert als neue Zelle im Zellen-Array der y-Werte hinzu (Index end+1)
- Speichern Sie die aktuell in die Datei geladenen Variablen.
2) Angenommen, die Datei mit diesem Namen existiert nicht:
- Erstellen Sie eine neue Datei mit dem in der Variablen "patientName" gespeicherten Namen
- Fügen Sie die aktuellen "DateandTime"-Daten als Zelle in das leere Zellenarray von x-Werten ein
- Fügen Sie die aktuellen "brainVolume"-Daten als Zelle in das leere Zellenarray von y-Werten ein
- Speichern Sie die aktuell in die Datei geladenen Variablen.
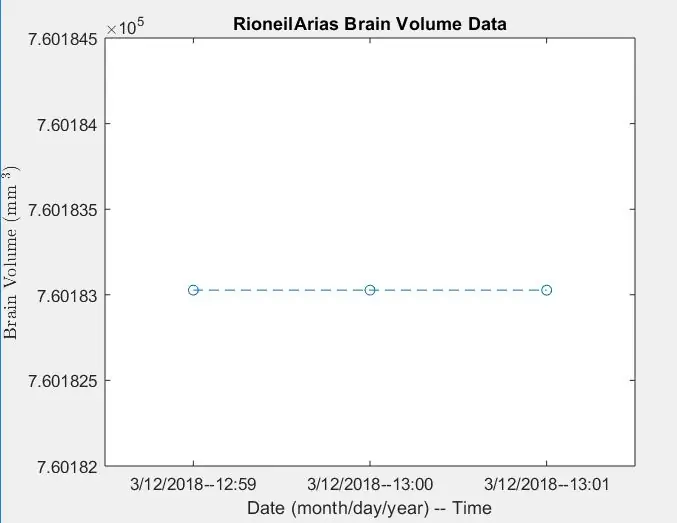
Schritt 12: Protokollieren von Daten: Anzeigen eines Diagramms des Gehirnvolumens über die Zeit

- Konvertieren Sie das x-Werte-Array (xVals) in ein kategoriales Array (xValsCategorical), um das Zeichnen zu ermöglichen
- Figurenfenster erstellen 5
- Zeichnen Sie die mit "xValsCategorical" und "yVals" bezeichneten Punkte (die das Gehirnvolumen enthalten), verwenden Sie Hohlkreise, um Punkte anzuzeigen und durch gestrichelte Linien zu verbinden
- Benennen Sie das Diagramm als: patientName Gehirnvolumendaten
- Beschriften Sie die x-Achse wie im Bild gezeigt
- Beschriften Sie die y-Achse wie im Bild gezeigt
- Sei Abbildung 5 gleich Ausgabe
Daraus ergibt die aufgerufene Funktion patientName eine Datei mit bearbeiteten Daten, die das Gehirnvolumen im Zeitverlauf verfolgen, und ein Diagramm, das Trends anzeigt.
Schritt 13: Lücken in Subplots schließen: Subplotclose.m

Die Funktion, angepasst an den Code von https://www.briandalessandro.com, dient dazu, die Lücken zwischen den Subplot-Figuren des Hauptcodes zu schließen, wenn die Figuren, die die MRT-Bilder und die Gehirnschichten anzeigen, erstellt werden. Die in subplotclose.m verwendete Subplot-Funktion passt die Position der gegebenen Subplots so an, dass sie im Aspekt der längeren Dimension eng aneinander passen. Wenn der Code beispielsweise eine 7 x 3-Matrix vorsieht, passen die Zeilen eng an, da die Zeilendimension länger ist. Wenn der Code eine 3 x 7-Matrix vorsieht, passen die Spalten eng an, mit Lücken in den Zeilen, wie in den Abbildungen unseres Hauptcodes gezeigt.
Schritt 14: Der Hauptcode: Alles löschen und Eingabeaufforderungen

Um den Hauptcode zu starten, klicken Sie auf die gleiche Schaltfläche mit der Aufschrift "Neu" in der oberen linken Ecke des Fensters und wählen Sie "Skript" anstelle von "Funktion" aus den vorherigen Abschnitten. Geben Sie den Code wie im Bild gezeigt im Editorfenster ein. Die Codezeilen erledigen die folgenden Aufgaben der Reihe nach:
- Schließen Sie alle geöffneten Dateien außer 0, 1 und 2.
- Schließen Sie alle Figurenfenster.
- Löschen Sie alle Variablen im Arbeitsbereich.
- Löschen Sie das Befehlsfenster.
- Anzeige im Befehlsfenster: Bitte geben Sie folgende Maße für die MRT-Aufnahmen ein:
- Fragen Sie in einer neuen Zeile im Befehlsfenster: Länge eines Scans in Millimetern:. Die vom Benutzer eingegebene Antwort wird auf die Variable "lengthMM" gesetzt.
- Fragen Sie in einer neuen Zeile: Abstand zwischen MRT-Scans in Millimetern:. Die vom Benutzer eingegebene Antwort wird auf die Variable "ZStacks" gesetzt.
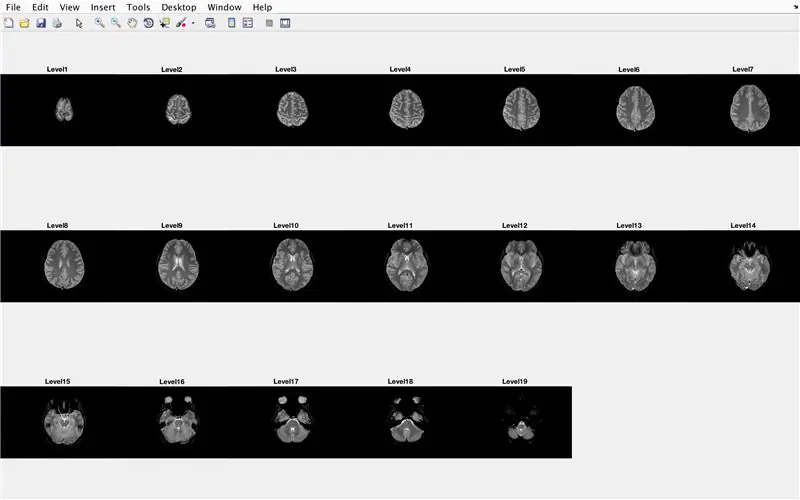
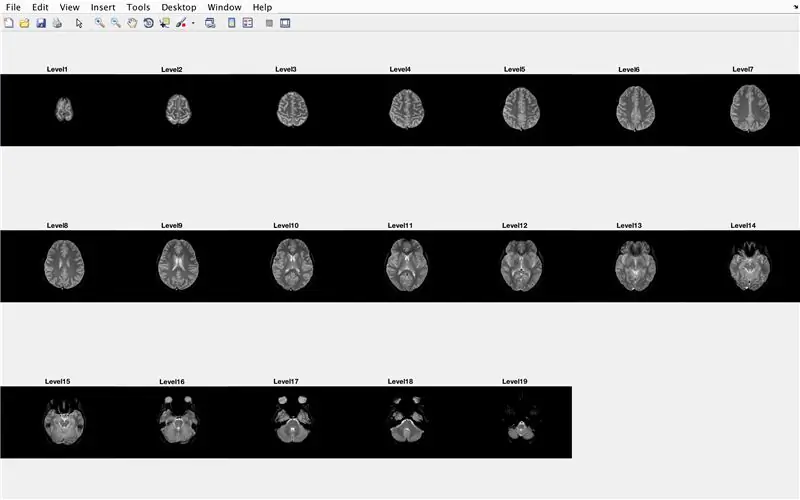
Schritt 15: Der Hauptcode: Stapelverarbeitung der Bilder

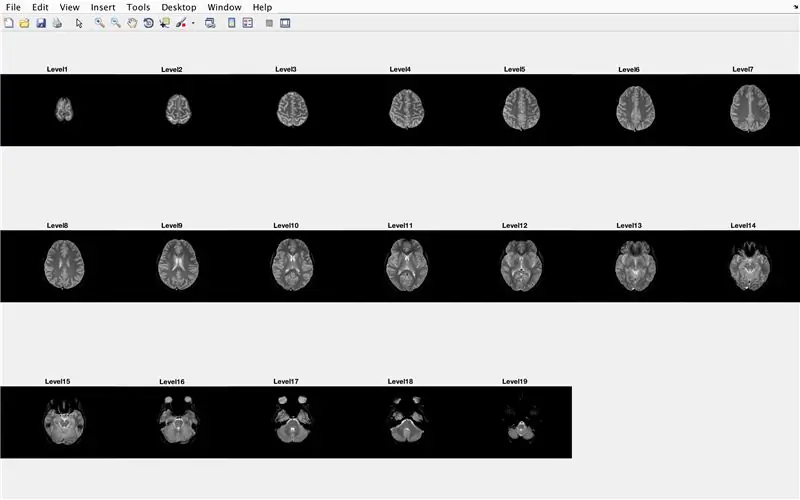
In diesem Abschnitt lädt der Code die Bilder (bestehend aus den MRT-Scans der Querschnitte des Gehirns) und speichert die Namen jeder Bilddatei in der Variablen "Base" und zeigt jeden der MRT-Scans an. Bitte folgen Sie dem Code im Bild, der Folgendes bewirkt:
- Erstellen Sie ein Strukturarray "BrainImages", das Informationen zu allen Dateien innerhalb des aktuellen Ordners enthält, die dem Namensformat von MRI_()-p.webp" />
- Variable "NumberofImages" gleich der Anzahl der Elemente im Strukturarray "BrainImages" setzen
- Figurenfenster öffnen 1
- Legen Sie eine for-Schleife fest, um die Anzahl der in der Datei gezählten Bilder zu durchlaufen
- Für jede Schleife ist "CurrentImage" der jeweilige Name jeder Datei MRI_i.png, mit der Iterationsnummer als 'i'
- Generieren Sie einen 3 x 7 Subplot, um die 19 Bilder anzuzeigen, die von "imshow" geladen werden sollen
- Zeigen Sie jedes Bild als weiteres Element im Subplot-Figurenfenster an
- Benennen Sie jedes Subplot-Element als Level_, wobei blank die Iterationsnummer der for-Schleife ist.
- Beenden der for-Schleife (Fehlervermeidung)
Dadurch werden im Abbildungsfenster 1 alle MRT-Scans in Rohform in einer 3 x 7-Konfiguration ohne Lücken in der x-Ausrichtung angezeigt.
Schritt 16: Der Hauptcode: Padding

Durch das Auffüllen vermeiden wir das Problem von geringfügigen Abweichungen in den Bildgrößen, die zu Fehlern bei Maßabweichungen führen können, wenn ein Bild etwas größer als ein anderes ist.
- Figurenfenster öffnen 2
- Laden Sie die Bildmatrix von MRI_1-p.webp" />
- Finden Sie die Größe der Bildmatrix und stellen Sie "OriginalXPixels" (für die Anzahl der Zeilen) und "OriginalYPixels" (für die Anzahl der Spalten) ein.
- Richten Sie die Matrix "BrainMat" so ein, dass sie aus allen Nullen besteht, mit 20 weiteren Zeilen und 20 weiteren Spalten für jede Ebene und 19 Gesamtquerschnitten, einer pro Ebene.
- Richten Sie "HolesMat" so ein, dass es aus demselben dreidimensionalen Array von Nullen besteht, um später die Lochkoordinaten einzugeben
- Erstellen Sie "zeroMat" mit der Größe von Pad plus zwanzig Zeilen und zwanzig Spalten, ein zweidimensionales Array von Nullen.
Schritt 17: Der Hauptcode: Grenzen bestimmen
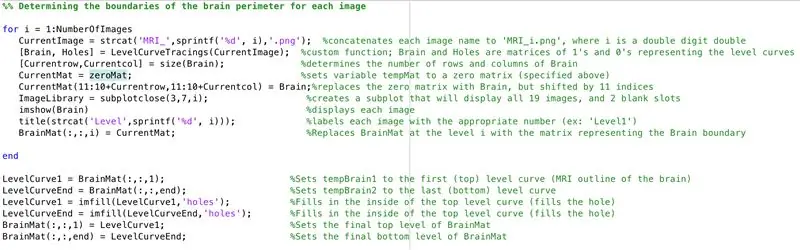
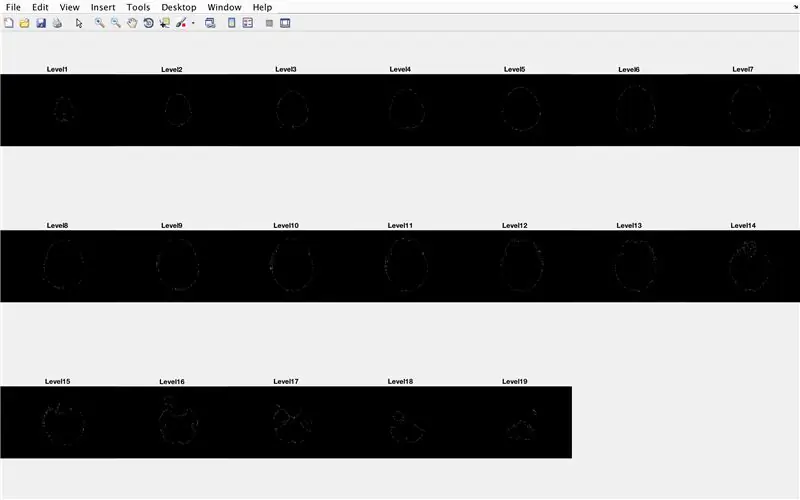
- Legen Sie eine for-Schleife fest, um die Daten von jedem zuvor geladenen Bild zu durchlaufen
- Auf die gleiche Weise wie bei der Stapelverarbeitung zuvor lädt "CurrentImage" Dateien mit "MRI_i.png", wobei i die Iterationsnummer ist
- Führen Sie jedes Bild durch die Verarbeitungsfunktion "LevelCurveTracings2.m", die Sie zuvor erstellt haben
- Ermitteln Sie die Größe der Ausgabe "Brain", setzen Sie die Anzahl der Zeilen auf "Currentrow" und die Anzahl der Spalten auf "Currentcolumns".
- Setzen Sie "CurrentMat" auf eine Null-Matrix mit den von "Currentrow" und "Currentcolumns" angegebenen Dimensionen
- Zentrieren Sie die Daten von "Brain" in "CurrentMat", mit einem Rand von 10 Zeilen auf allen Seiten
- Generieren Sie ein Subplot mit den Maßen 3 x 7, um die Grenzen der Bilder anzuzeigen
- Benennen Sie jedes der Unterplotelemente im Figurenfenster
- Generieren Sie eine dreidimensionale Matrix "BrainMat", die aus jeder Begrenzungsschicht "CurrentMat" besteht
- Beenden der for-Schleife (um Fehler zu vermeiden)
Der folgende Unterabschnitt füllt die Löcher oben und unten in der vorgeschlagenen dreidimensionalen Form aus
- Setze "LevelCurve1" gleich der ersten Ebene von "BrainMat" (Unterseite des Volumenkörpers)
- Setze "LevelCurveEnd" gleich der letzten Ebene von "BrainMat" (Oberseite des Volumenkörpers)
- Überschreibe "LevelCurve1" mit einer ausgefüllten Ebene
- Überschreibe "LevelCurveEnd" mit einer ausgefüllten Ebene
- Legen Sie die ausgefüllte Ebene als unterste Ebene von "BrainMat" fest
- Legen Sie die ausgefüllte Ebene als oberste Ebene von "BrainMat" fest
Schritt 18: Der Hauptcode: Bestimmen des richtigen Z-Maßes

Die ersten drei Zeilen bestehen aus dem Einrichten eines leeren Arrays "z" und dem Ausführen einfacher Konvertierungsoperationen (Teilen der Pixel durch die Länge), um das Volumen in mm^3. richtig abzulesen
- Erstellen Sie eine for-Schleife, um durch jede Ebene zu blättern
- Ermitteln Sie die Anzahl der Einsen in einer bestimmten Schicht
- Konvertieren Sie die z-Koordinaten für die Einsen in Werte, die auf das richtige Verhältnis skaliert sind, setzen Sie auf "tempz", einen Spaltenvektor
- Addiere z-Wert für die Niveaukurve zum Vektor z
Damit sind die z-Koordinaten richtig eingestellt.
Schritt 19: Der Hauptcode: Bestimmung der X- und Y-Koordinaten

Bestimmen Sie nun die x- und y-Positionen jedes der Punkte in den Grenzen.
- Initialisiere "xBrain" als leeres Array
- Initialisiere "yBrain" als leeres Array
- Richten Sie eine for-Schleife ein, um jedes geladene Bild zu durchlaufen
- Kompilieren Sie eine zweispaltige Matrix, um die planaren Koordinaten jedes Punktes in der Grenze zu speichern, dargestellt durch die Spaltenvektoren "RowBrain" und "ColumnBrain"
- Hängen Sie "xBrain" mit den aktuell gefundenen "RowBrain"-Koordinaten an
- Hängen Sie "yBrain" mit den aktuell gefundenen "ColumnBrain"-Koordinaten an
- Beenden der for-Schleife (um Fehler zu vermeiden)
Schritt 20: Der Hauptcode: Zeichnen einer dreidimensionalen Struktur, Finden des Volumens und Protokollieren von Daten
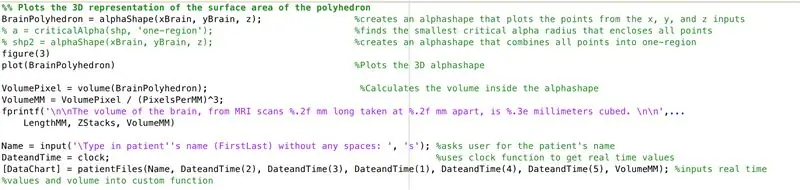

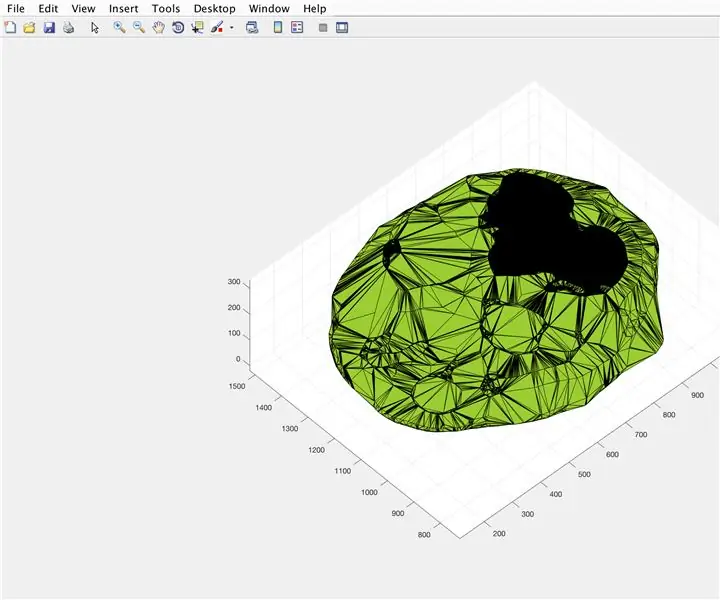
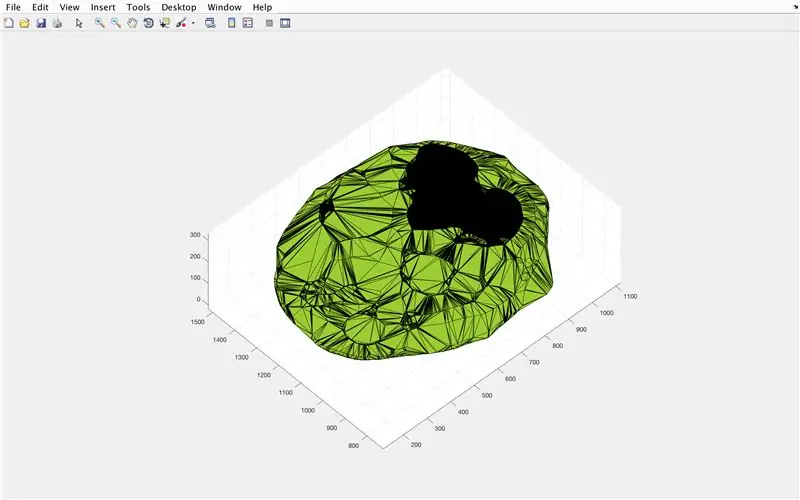
Mit der Funktion alphaShape erstellen wir eine dreidimensionale Struktur, aus der wir das Volumen des Gehirns berechnen können.
- Verwenden Sie die Funktion alphaShape, fügen Sie die Vektoren "xBrain", "yBrain" und "z" für die x-, y- und z-Koordinaten ein und setzen Sie sie gleich "BrainPolyhedron"
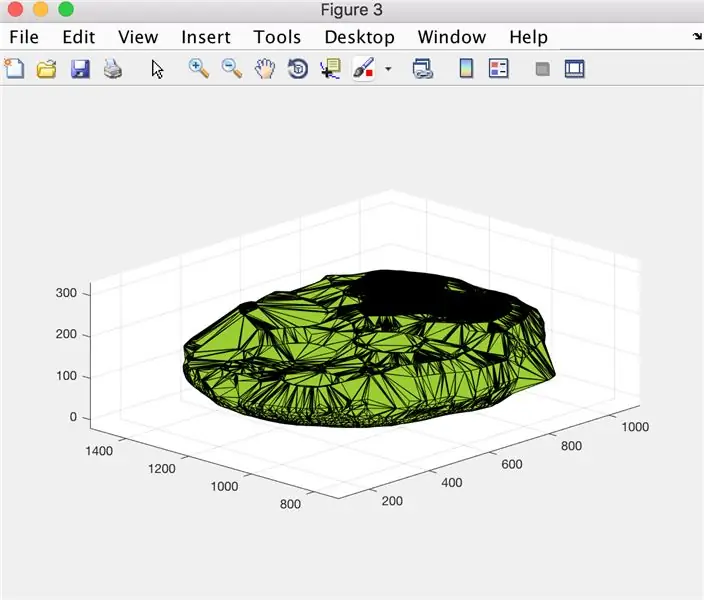
- Figurenfenster öffnen 3
- Plotten Sie die berechnete Alphaform "BrainPolyhedron", Anzeige im Figurenfenster
- Berechnen Sie das Volumen der Alphaform mit einer Funktion "Volumen", die für Alphaformen funktioniert
- Konvertieren Sie das Volumen in mm^3
- Drucken Sie das Volumen des Volumenkörpers im Befehlsfenster
- Aufforderung zur Eingabe eines Patientennamens als Eingabe
- Holen Sie sich das aktuelle Datum und die aktuelle Uhrzeit mit der Uhr und stellen Sie "DateandTime" ein
- Rufen Sie die Funktion "patientFiles" auf, um die berechneten Daten zu protokollieren und zu plotten
Von hier aus sollten das zweite und dritte Bild die Figuren zeigen, die auftauchen, und das vierte Bild das, was im Befehlsfenster angezeigt werden soll.
Empfohlen:
DIY So zeigen Sie die Zeit auf dem M5StickC ESP32 mit Visuino an - Einfach zu tun: 9 Schritte

DIY So zeigen Sie die Zeit auf dem M5StickC ESP32 mit Visuino an - Einfach zu machen: In diesem Tutorial lernen wir, wie Sie den ESP32 M5Stack StickC mit Arduino IDE und Visuino programmieren, um die Zeit auf dem LCD anzuzeigen
So starten Sie Ihren Computer jeden Tag oder zu jeder Zeit automatisch – wikiHow

So starten Sie Ihren Computer automatisch jeden Tag oder wann immer: In dieser Anleitung erfahren Sie, wie Sie Ihren Computer jeden Tag zu einer bestimmten Zeit automatisch starten lassen. Dies ist auch meine erste Anleitung
Zeit zu leben: 5 Schritte

Time to Live: Wir kombinieren die Magie, alle Harry-Potter-Filme in einer Woche zu sehen, und den Networking-Begriff TTL (Time To Live)
So verfolgen Sie Bilder in Photoshop Elements 6: 6 Schritte

So verfolgen Sie Bilder in Photoshop Elements 6: In diesem anweisbaren zeige ich Ihnen, wie Sie jedes Bild verfolgen und es so aussehen lassen, wie Sie es skizziert haben. Dies ist ziemlich einfach und wenn Sie möchten, können Sie es detaillierter machen. Dazu benötigen Sie: 1. Photoshop Elements 6 (Oder jede Form von Photoshop
WIE MAN EINE REPLIK DER BERÜHMTEN ALTOIDS ZINN HERSTELLT. (UND ZUR GLEICHEN ZEIT RECYCELN): 7 Schritte

WIE MAN EINE REPLIK DER BERÜHMTEN ALTOIDS ZINN HERSTELLT. (UND ZUR GLEICHEN ZEIT RECYCELN): Hi, bei diesem unzerstörbaren Gegenstand geht es darum, wie man eine Metallbox in der gleichen Größe (oder der gewünschten Größe) der Altoids-Dose herstellt. WISSEN SIE, WAS DAS BEDEUTET????Das bedeutet, dass Sie Ihre eigenen Dosen herstellen können….. JEDER GRÖSSE, DIE SIE WOLLEN!!!!!!!!!!!So geht's.MATERIALIEN: 2
