Inhaltsverzeichnis:
- Schritt 1: Voraussetzungen
- Schritt 2: Voraussetzungen (Fortsetzung)
- Schritt 3: Voraussetzungen (Fortsetzung)
- Schritt 4: Voraussetzungen (Fortsetzung)
- Schritt 5: Bereinigen Sie Matlab, um das Ausführen von Code vorzubereiten
- Schritt 6: Wählen Sie 10 normale Augenbilder und 10 Bilder mit diabetischen Retinopathie-Symptomen
- Schritt 7: Wählen Sie 10 normale Augenbilder und 10 Bilder mit Symptomen der diabetischen Retinopathie (Fortsetzung)
- Schritt 8: Erstellen Sie 2 Variablen (normal und diagnostiziert) und setzen Sie sie jeweils gleich 0
- Schritt 9: Erstellen Sie eine for-Schleife, um normale Bilder automatisch hochzuladen
- Schritt 10: Erstellen Sie eine for-Schleife, um normale Bilder automatisch hochzuladen (Fortsetzung)
- Schritt 11: Beschneiden Sie die Ränder des Bildes
- Schritt 12: Erstellen Sie ein Graustufenbild
- Schritt 13: Erstellen Sie ein kontrastreiches Bild
- Schritt 14: Verbessern Sie das Kontrastbild
- Schritt 15: Erstellen Sie einen Durchschnittsfilter
- Schritt 16: Kombinieren Sie den Mittelungsfilter mit dem kontrastierten Bild
- Schritt 17: Erstellen Sie eine neue mittlere Maske durch Subtrahieren von Pixeln
- Schritt 18: Erstellen Sie ein binär gefiltertes Bild
- Schritt 19: Entfernen Sie kleinere Blobs, die in den gefilterten Bildern gefunden wurden
- Schritt 20: Erstellen Sie ein Plattenstrukturierungselement
- Schritt 21: Morphologische Abschlussoperationen durchführen
- Schritt 22: Finden Sie die Objekte mit einer Konnektivität von mindestens 8
- Schritt 23: Finden Sie die maximale Anzahl verbundener Pixel
- Schritt 24: Setzen Sie die maximalen Pixelwerte auf 0 und finden Sie Pixel mit >=26 Pixel Connectivity
- Schritt 25: Blutgefäße im Bild entfernen
- Schritt 26: Figurenanzeige
- Schritt 27: Gefäße entfernen und Blutklumpen zählen
- Schritt 28: Diagnostizieren Sie das Netzhautbild basierend auf der Anzahl der identifizierten Blutgerinnsel
- Schritt 29: Wenn mehr als 5 Blobs vorhanden sind …
- Schritt 30: Wiederholen Sie den Filtervorgang für normale Bilder mit Bildziffernwerten wie 2 und 3
- Schritt 31: Wiederholen Sie den gesamten Vorgang für die diagnostizierten Bilder
- Schritt 32: Statistische Analyse
- Schritt 33: Konfidenzintervall finden
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 07:21.
- Zuletzt bearbeitet 2025-01-23 12:52.
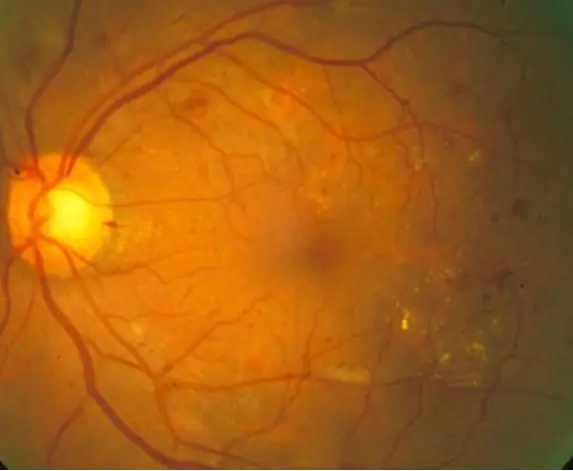

(Siehe Codeübersicht oben)
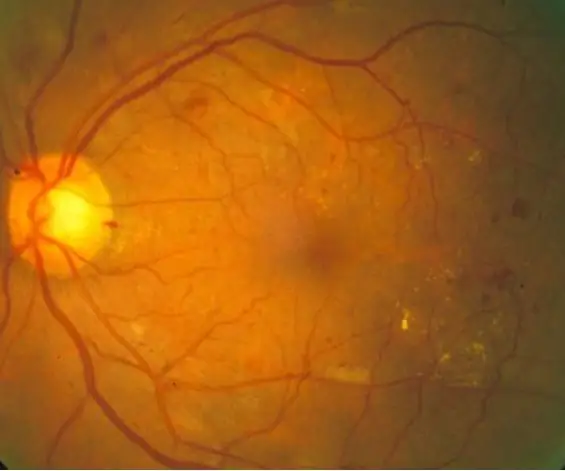
Die diabetische Retinopathie ist eine diabetische Augenerkrankung, die durch einen hohen Blutzuckerspiegel verursacht wird. Durch den hohen Blutzuckerspiegel schwellen die Blutgefäße in der Netzhaut an, was zu vergrößerten Blutgefäßen und sogar Gefäßlecks führt, die zu dunklen Flecken in Netzhautbildern führen. Mit diesem Code möchten wir das Auftreten von Blutgefäßleckstellen als Indikator für eine diabetische Retinopathie im Hintergrund verwenden, obwohl in der realen Welt weitere Diagnosetechniken erforderlich wären. Das Ziel dieses Codes ist es, die Bildverarbeitung und Diagnose von Netzhautbildern zu automatisieren, um Anzeichen einer diabetischen Retinopathie zu erkennen, die sich durch dunkle Flecken in den Netzhautbildern zeigen.
10 normale Netzhautbilder und 10 diagnostizierte Netzhautbilder wurden durch einen Code verarbeitet, der zuerst die Bilder liest und filtert und dann die dunklen Flecken quantifiziert, um zu bestimmen, ob diabetische Retinopathie-Symptome vorhanden sind, basierend auf einem gegebenen Schwellenwert. Die Ergebnisse werden dann zur Interpretation durch den Betrachter auf das Befehlsfenster gedruckt.
Schritt 1: Voraussetzungen
1. Stellen Sie sicher, dass Sie das MATLAB-Programm auf Ihren Computer heruntergeladen haben.
2. Laden Sie die im Link gefundene txt-Datei herunter. (Drücken Sie 'Strg+s', um im selben Verzeichnis wie den MATLAB-Code zu speichern)
Schritt 2: Voraussetzungen (Fortsetzung)
4. Öffnen Sie MATLAB und geben Sie ‚uiimport‘in das Befehlsfenster ein.
5. Wählen Sie die Datei Officialdiagnoses.txt aus und importieren Sie sie als Zellmatrix in MATLAB.
6. Stellen Sie sicher, dass im Arbeitsbereich „offizielle Diagnosen“als Variable angezeigt werden.
Schritt 3: Voraussetzungen (Fortsetzung)

7. Laden Sie die Funktion ModWald.m herunter, die Sie aus dem obigen Code erhalten können, oder laden Sie sie von Canvas herunter.
(Code bereitgestellt von Professor King und Professor Choi)
Schritt 4: Voraussetzungen (Fortsetzung)

8. Laden Sie die 400 Rohbilder aus dem Datenbereich des STARE-Projekts herunter.
Schritt 5: Bereinigen Sie Matlab, um das Ausführen von Code vorzubereiten

Zum Code hinzufügen:
1. Alle schließen (Schließt alle zuvor geöffneten Bilder)
2. clearvars - außer offiziellen Diagnosen (Löscht alle Variablen außer der zuvor importierten offiziellen Diagnose-txt-Datei)
3. cclc (Befehlsfenster löschen)
Schritt 6: Wählen Sie 10 normale Augenbilder und 10 Bilder mit diabetischen Retinopathie-Symptomen
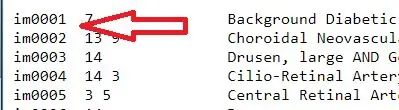
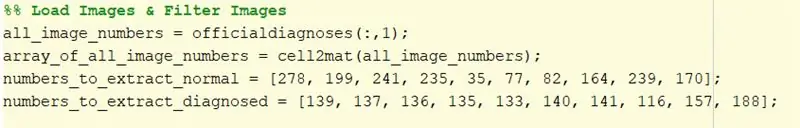
1. Nehmen Sie die Diagnosetextdatei und extrahieren Sie die Bildnamen. Diese Namen sind in der ersten Spalte der Textdatei enthalten. Um sie zu extrahieren, geben Sie 'officialdiagnoses(:, 1)' ein. Die Matrix der Bildnamen wurde einer Variablen „all_image_numbers“zugewiesen.
2. Konvertieren Sie die Variable all_image_numbers von einem Zellen-Array in ein Matrix-Array mit der Funktion cell2mat
Schritt 7: Wählen Sie 10 normale Augenbilder und 10 Bilder mit Symptomen der diabetischen Retinopathie (Fortsetzung)
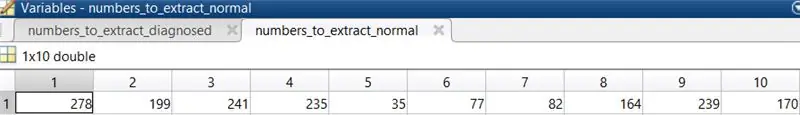
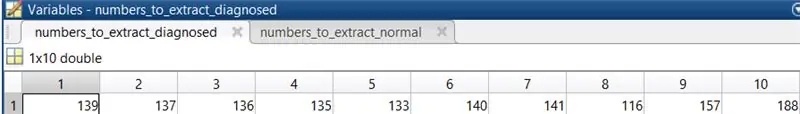
3. Wählen Sie 10 normale Augenbilder aus, um den Code auszuführen. Die ausgewählten Bilder waren in diesem Fall 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Platzieren Sie diese Zahlen in einer Matrix und weisen Sie sie einer Variablen zu, die beim Laden der Bilder aufgerufen wird.
4. Wiederholen Sie Schritt 3 für Netzhautbilder, bei denen diabetische Retinopathie diagnostiziert wurde. Die ausgewählten Bilder waren in diesem Fall 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Schritt 8: Erstellen Sie 2 Variablen (normal und diagnostiziert) und setzen Sie sie jeweils gleich 0

Erstellen Sie diese Variablen vor der for-Schleife, um die Schleifennummern zu initialisieren.
Schritt 9: Erstellen Sie eine for-Schleife, um normale Bilder automatisch hochzuladen

1. Erstellen Sie eine for-Schleife
2. Setzen Sie eine Zählvariable (in diesem Fall i) auf eine Matrix mit den Werten 1-10. Diese Zählvariable wird verwendet, um jedes Bild einzeln aufzurufen
3. Nehmen Sie das i-Element in der Bildermatrix, um den Bildnamen zu extrahieren und mit der Funktion num2str aus einer Zeichenfolge in eine Zahl umzuwandeln.
Ermitteln Sie die Anzahl der Ziffern im Bildnamen mit der Numel-Funktion. Weisen Sie diesen Wert einer Variablen zu, digits_normal. Diese Zahl sollte 1 für einstellige Zahlen, 2 für zweistellige Zahlen und 3 für dreistellige Zahlen sein. Diese Informationen werden verwendet, um Bilder automatisch aufzurufen.
Schritt 10: Erstellen Sie eine for-Schleife, um normale Bilder automatisch hochzuladen (Fortsetzung)

3. Erstellen Sie eine if-Anweisung, die alle drei Möglichkeiten aus den vorherigen Schritten enthält. Wenn der Bildname 1-stellig ist, wird das Bild als „im000“bezeichnet, wenn er 2-stellig ist, wird das Bild als „im00“bezeichnet und wenn er 3 hat, wird das Bild als „im0“bezeichnet.
4. Weisen Sie unter jeder if-Anweisung eine Variable zu, um „im“unter der entsprechenden if-Anweisung mit der entsprechenden Anzahl von Nullen (wie oben beschrieben) zu lesen, gefolgt von i.
Schritt 11: Beschneiden Sie die Ränder des Bildes
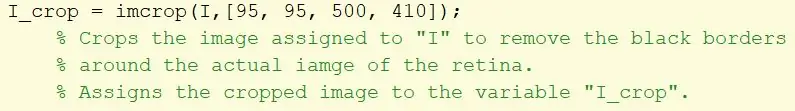
Nehmen Sie das Originalbild und wenden Sie einen Imcrop-Filter an, um schwarze Ränder zu beseitigen, und weisen Sie es einer Variablen I_crop zu. Das Zuschneiderechteck wird mit einer Matrix [95, 95, 500, 410] angegeben.
Schritt 12: Erstellen Sie ein Graustufenbild

Nehmen Sie das zugeschnittene Bild und wenden Sie den rbg2gray-Filter an, um das Bild in Graustufen zu ändern. Weisen Sie dieses Bild der Variablen I2 zu.
Schritt 13: Erstellen Sie ein kontrastreiches Bild
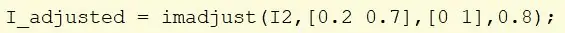
Nehmen Sie das Bild I2 und verwenden Sie Imadjust, um die Intensitätswerte neu zu skalieren.
Nehmen Sie Werte, die in den Bereich [0,2, 0,7] fallen, und skalieren Sie sie auf [0, 1]. Das Gamma wird auf 0,8 eingestellt, um das Bild heller zu machen. Weisen Sie das neue Bild I_adjusted zu.
Schritt 14: Verbessern Sie das Kontrastbild
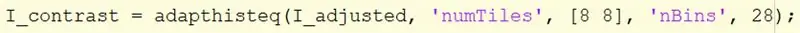
Nehmen Sie das I_adjusted-Bild und verwenden Sie die Funktion adapthisteq, um den Kontrast zu verbessern.
Die Adapthisteq-Syntax erfordert den Bildnamen I_adjusted, „numTiles“, die Größe von numTiles, „nBins“und die Anzahl der Bins. Die Größe von numTiles wird auf [8 8] gesetzt, wodurch das Bild in 8x8 Kacheln unterteilt wird, und die Anzahl der Bins wird auf 28 gesetzt. Weisen Sie das Bild I_constrast zu.
Schritt 15: Erstellen Sie einen Durchschnittsfilter

Erstellen Sie mit der Funktion fspecial eine Variable namens 'meanfilt'. Geben Sie „Durchschnittsfunktion“ein, um den Mittelungsfilter zu erstellen, und fügen Sie [90 90] für die gleitende Fenstergröße ein.
Schritt 16: Kombinieren Sie den Mittelungsfilter mit dem kontrastierten Bild
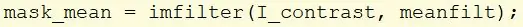
Erstellen Sie eine neue Variable namens mask_mean und verwenden Sie die imfilter-Funktion, um das I_contrast-Bild aufzunehmen und den zuvor erstellten Mittelwertfilter anzuwenden.
Schritt 17: Erstellen Sie eine neue mittlere Maske durch Subtrahieren von Pixeln

Erstellen Sie eine Variable namens mask_mean2 und verwenden Sie die Funktion imsubtract, um den Wert jedes Pixels in I_contrast vom entsprechenden Pixel in mask_mean zu subtrahieren.
Schritt 18: Erstellen Sie ein binär gefiltertes Bild

Verwandeln Sie Graustufenbilder mit Imbinarisieren in Schwarzweiß. Eingabe mask_mean2, ‚adaptive‘, ‚ForegroundPolarity‘, ‚dark‘, ‚Sensitivity‘, 0.6. Weisen Sie dieses neue Bild mask_binarize zu.
Schritt 19: Entfernen Sie kleinere Blobs, die in den gefilterten Bildern gefunden wurden
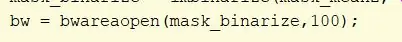
Entfernen Sie Objekte mit einer Konnektivität von weniger als 100 Pixeln mithilfe der bwareaopen-Funktion auf mask_binarize und setzen Sie den Schwellenwert auf 100. Weisen Sie die Variable als bw zu.
Schritt 20: Erstellen Sie ein Plattenstrukturierungselement

Erstellen Sie ein Scheibenstrukturierungselement (mit einem Radius von 2) mit der strel-Funktion. Weisen Sie es se zu.
Schritt 21: Morphologische Abschlussoperationen durchführen

Nehmen Sie bw und wenden Sie die imclose-Funktion auf das Strukturelement an, um eine morphologische Close-Operation am Objekt durchzuführen.
Schritt 22: Finden Sie die Objekte mit einer Konnektivität von mindestens 8
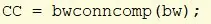
Nehmen Sie bw und verwenden Sie bwconncomp, um die Objekte mit einer Konnektivität von mindestens 8 im Bild zu finden. Weisen Sie die Nummernausgabe cc_1 zu.
Schritt 23: Finden Sie die maximale Anzahl verbundener Pixel
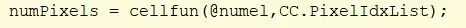
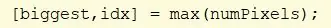
Verwenden Sie die cellfun-Funktion, um die Funktion „numel“für jede Zelle in CC auszuführen. Dadurch wird die Anzahl der Elemente in der Zelle PixelIdxList ermittelt. Weisen Sie „numPixels“einen Wert zu.
Finden Sie die Maximalwerte in numPIxels. Weisen Sie „biggest“das größte Maximum und „idx“den Index des Maximalwerts zu.
Schritt 24: Setzen Sie die maximalen Pixelwerte auf 0 und finden Sie Pixel mit >=26 Pixel Connectivity
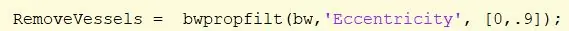



Setzen Sie die Pixel mit den größten Werten im Bild „bw“auf 0, wodurch die Pixel schwarz werden.
Suchen Sie mit bwconncomp die Objekte mit einer Konnektivität von mindestens 26 Pixeln im Bild. Zu Variable cc_1 zuweisen.
Schritt 25: Blutgefäße im Bild entfernen
Entfernen Sie die noch im Bild vorhandenen Blutgefäße mit der bwpropfilt-Funktion mit einem Bereich von [0, 0,9].
[0.9, 1] ist ausgeschlossen, da Werte nahe 1 eine Linie anzeigen. Weisen Sie „Gefässe entfernen“zu.
Schritt 26: Figurenanzeige
Zeigen Sie jedes gefilterte Bild in einem Subplot an. Imshow. mit „border“- und „tight“-Eingaben, zeigt jedes Bild in einer Subplot-Struktur an. Fügen Sie jedem Bild einen Titel hinzu, um zu unterscheiden, welcher Filter verwendet wurde.
Schritt 27: Gefäße entfernen und Blutklumpen zählen
1. Nehmen Sie „RemoveVessels“und wenden Sie die „Centroid“-Funktion in Regionprops an, um die Schwerpunkte der Objekte im Bild zu identifizieren. Diese Objekte sollten den im Bild vorhandenen Blutgerinnseln entsprechen.
2. Zählen Sie die Anzahl der identifizierten Blutgerinnsel, indem Sie die Länge der Schwerpunktmatrix nehmen.
Schritt 28: Diagnostizieren Sie das Netzhautbild basierend auf der Anzahl der identifizierten Blutgerinnsel
Verwenden Sie if-Anweisungen, um das Bild basierend auf der Anzahl der identifizierten Blutgerinnsel zu diagnostizieren.
Wenn die Anzahl der identifizierten Schwerpunkte kleiner oder gleich 5 war, wurde das Bild als normal identifiziert.
Wenn die Anzahl der Zentroide größer als 5 war, wurde das Bild mit diabetischer Retinopathie diagnostiziert.
Das Ergebnis wird mit fprintf in das Befehlsfenster gedruckt.
Schritt 29: Wenn mehr als 5 Blobs vorhanden sind …
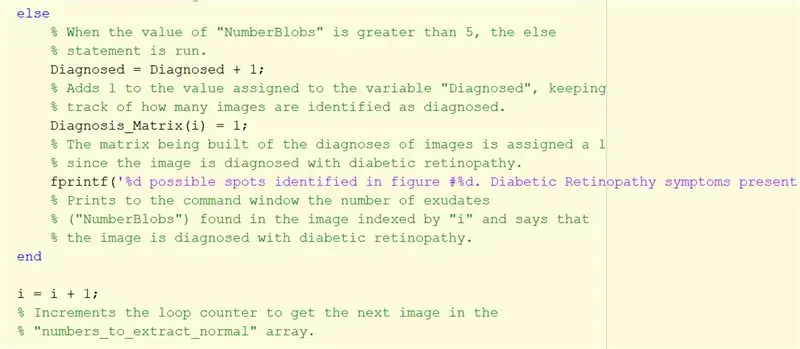
Wiederholen Sie die obigen Anweisungen für diagnostizierte Bilder als eine else-Anweisung. Dieser Teil wird ausgeführt, wenn die Anzahl der Blobs größer als 5 ist.
Beenden Sie die if-Anweisung.
Schritt 30: Wiederholen Sie den Filtervorgang für normale Bilder mit Bildziffernwerten wie 2 und 3
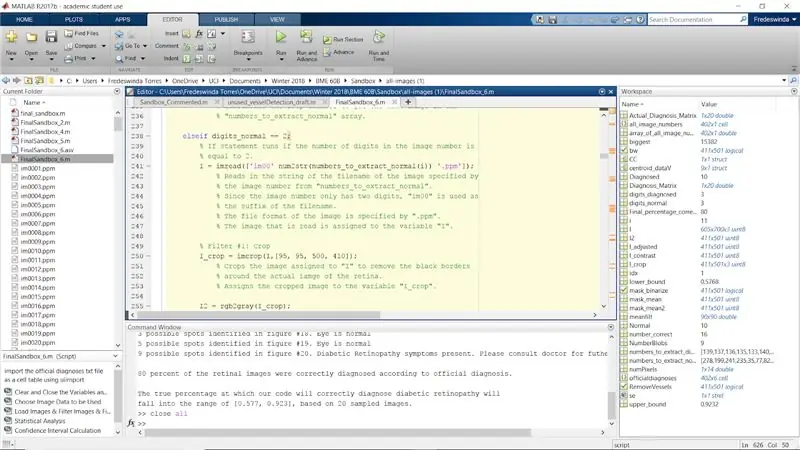
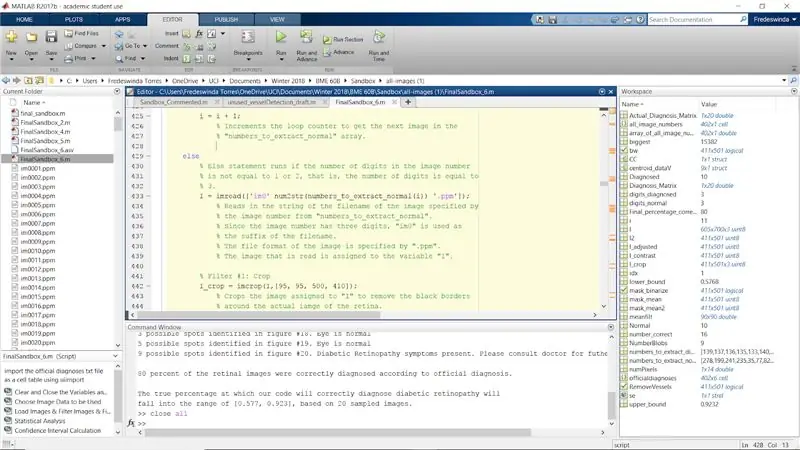
Wiederholen Sie den Vorgang für den Rest der ursprünglichen if-Anweisungen, wenn numel (die Anzahl der Stellen in der Bildnummer) gleich 2 und 3 ist. Damit ist die for-Schleife für die normalen Bilder abgeschlossen.
Beende die for-Schleife.
Schritt 31: Wiederholen Sie den gesamten Vorgang für die diagnostizierten Bilder
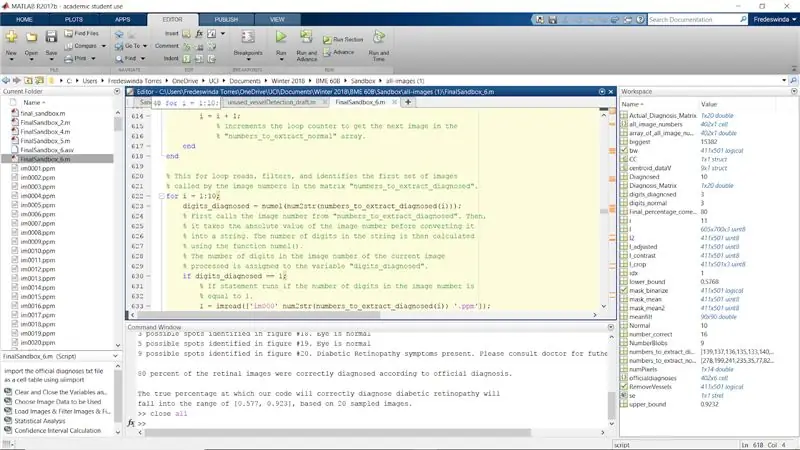
Wiederholen Sie den gesamten Vorgang mit den durch die Matrix „numbers_to_extract_diagnosed“aufgelisteten diagnostizierten Bildern.
Stellen Sie sicher, dass Sie jede Zahl (i) durchgehen und in Zahl (i + 10) ändern, damit die diagnostizierten Zahlen als Bilder 11 bis 20 angezeigt werden.
Schritt 32: Statistische Analyse
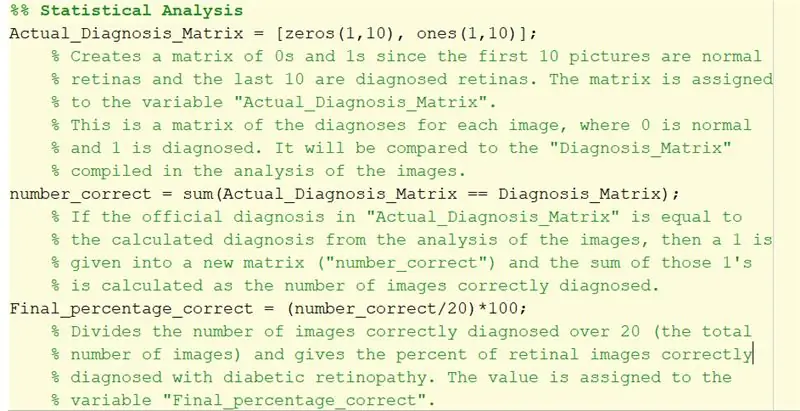
1. 'Actual_Diagnosis_Matrix' wird verwendet, um die Ergebnisse mit der offiziellen Diagnose in der txt-Datei zu vergleichen. Die ersten 10 Nullen zeigen an, dass die ersten 10 Bilder normal sein sollten. Die letzten 10 zeigen an, dass die letzten 10 Bilder als diabetische Retinopathie klassifiziert werden sollten.
2. Das doppelte Gleichheitszeichen, das verwendet wird, um 'number_correct' zu erstellen, erstellt ein logisches Array, indem es den Wert der entsprechenden Elemente von 'Actual_Diagnosis_Matrix' mit 'Diagnosis_Matrix' vergleicht, die aus der for-Schleife erstellt wurden.
Für jedes Element, das mit der Diagnose übereinstimmt, wird eine 1 hinzugefügt, was bedeutet, dass der Code dieses Bild korrekt diagnostiziert hat. Wenn es falsch ist, wird der Matrix eine 0 hinzugefügt.
Dann addiert man die Summe aller Einsen. Mit anderen Worten, es findet die Summe der richtig diagnostizierten Bilder.
3. 'Final_percentage_correct' ist der berechnete Prozentsatz der Genauigkeit des Codes der diagnostizierten diabetischen Retinopathie. Die Anzahl der richtig diagnostizierten Bilder wird durch 20 (die Gesamtzahl der Bilder) geteilt und mit 100 multipliziert, um den Prozentsatz der erfolgreichen Diagnosen zu ermitteln.
Schritt 33: Konfidenzintervall finden
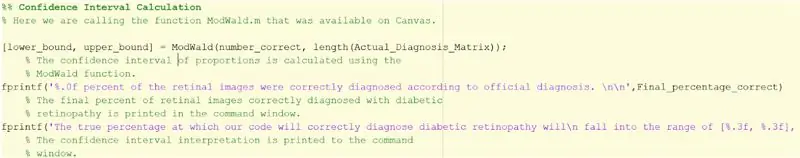
1. Stellen Sie sicher, dass Sie ModWald.m heruntergeladen haben, um es als Funktion aufzurufen. Ohne die Funktion müssten Sie das Konfidenzintervall mit der modifizierten Wald-Methode selbst berechnen.
2. Die ModWald-Funktion hat 2 Eingänge, wobei der erste die Anzahl der korrekt identifizierten Bilder und der zweite die Gesamtzahl der Bilder ist.
3. Die ModWald-Funktion gibt die untere und obere Grenze des Konfidenzintervalls der Proportionen für die Genauigkeit der abgetasteten Daten aus. Mit anderen Worten, Sie geben ein Prozentintervall an, in dem der wahre Prozentsatz der Genauigkeit des Codes liegt.
4. Verwenden Sie unten fprintf, um die Statistik und das Konfidenzintervall im Befehlsfenster auszugeben.
> fprintf('%.0f Prozent der Netzhautbilder wurden laut offizieller Diagnose richtig diagnostiziert. \n\n', Final_percentage_correct)
> fprintf('Der wahre Prozentsatz, bei dem unser Code diabetische Retinopathie korrekt diagnostiziert, wird\n in den Bereich von [%.3f, %.3f] fallen, basierend auf 20 abgetasteten Bildern \n', untere_Grenze, obere_Grenze)
Empfohlen:
Einen Stromzähler über Arduino auslesen – wikiHow

So lesen Sie einen Stromzähler über Arduino aus: Oft wäre es interessant, den aktuellen Stromverbrauch oder den Gesamtstromverbrauch Ihres Hauses zu kennen, um Ihre Stromkosten zu begrenzen und die Umwelt zu schonen. Das ist kein wirkliches Problem, denn meistens findet man ein smartes digitales el
So erstellen Sie einen Smart Pot mit NodeMCU, der von der App gesteuert wird – wikiHow

So erstellen Sie einen Smart Pot mit NodeMCU, der von einer App gesteuert wird: In dieser Anleitung bauen wir einen Smart Pot, der von einem ESP32 und einer Anwendung für Smartphones (iOS und Android) gesteuert wird. Wir verwenden NodeMCU (ESP32) für die Konnektivität und die Blynk-Bibliothek für das Cloud-IoT und die Anwendung auf dem Smartphone. Endlich haben wir
Arduino-Programmierung über das Handy -- Arduinodroid -- Arduino-Ide für Android -- Blinzeln: 4 Schritte

Arduino-Programmierung über das Handy || Arduinodroid || Arduino-Ide für Android || Blink: Bitte abonnieren Sie meinen Youtube-Kanal für weitere Videos…… Arduino ist ein Board, das direkt über USB programmiert werden kann. Es ist sehr einfach und günstig für College- und Schulprojekte oder sogar für Produktprototypen. Viele Produkte bauen zunächst darauf für i
Steuern Sie Geräte über das Internet über den Browser. (IoT): 6 Schritte

Steuern Sie Geräte über das Internet über den Browser. (IoT): In diesem Instructable zeige ich Ihnen, wie Sie Geräte wie LEDs, Relais, Motoren usw. über das Internet über einen Webbrowser steuern können. Und Sie können über jedes Gerät sicher auf die Bedienelemente zugreifen. Die Webplattform, die ich hier verwendet habe, ist RemoteMe.org visit
Inventar - Schnelle PC-Diagnose jetzt abgelöst, siehe unten: 6 Schritte

Inventar - Schnelle PC-Diagnose jetzt abgelöst, siehe unten: Wenn Sie jemals an einem PC arbeiten müssen, der sich nicht vor Ihnen befindet, benötigen Sie eine genaue Inventarisierung. Dieses instructable kam rein, weil ein Kollege Schwierigkeiten hat, einen Treiber für einen PC zu finden. Ich werde Aida32 Personal von http://majorgeeks.com verwenden
