Inhaltsverzeichnis:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 07:19.
- Zuletzt bearbeitet 2025-01-23 12:52.
Von: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Dieser MATLAB-Code ist ein Programm zur Erkennung der genauen Größe, Form und Lage eines Tumors, der in MRT-Scans des Gehirns eines Patienten gefunden wurde. Dieses Programm wurde ursprünglich für die Tumorerkennung bei MRT-Untersuchungen des Gehirns entwickelt, kann aber auch für die Krebsdiagnostik bei anderen Organscans verwendet werden.
Die folgenden Anweisungen beschreiben zunächst die Methoden zur Bildanalyse durch Filtern und Bereinigen des MRT-Scans, durch Binarisierung, Medianfilterung und Schiebefenster. Als nächstes wird es Anweisungen geben, wie der Tumor unter Verwendung einer vorgenerierten elliptischen Maske isoliert und weiter gefiltert wird, um den Umfang der Form des Tumors zu umreißen.
Sobald der Tumor erkannt wurde, wird in den Anweisungen weiter beschrieben, wie dieses Programm in eine grafische Benutzeroberfläche (GUI) integriert wird. In dieser Anleitung werden der entsprechende Code und die entsprechenden Dateien angehängt, um zu erklären, wie diese MRT-Scan-Analyse funktioniert.
Einige Dinge zu wissen, herunterzuladen und bereit zu haben, bevor Sie mit diesem instructable fortfahren: 1. Stellen Sie sicher, dass Sie die neueste Version von MATLAB heruntergeladen haben. Sie können R2018b hier installieren:
2. Um dieses Programm auszuführen, benötigen Sie Zugriff auf MRT-Gehirnscandateien. Obwohl einige immer aus Google-Bildern zu finden sind, kann eine gründliche und genaue Analyse anhand der richtigen Bilder verschiedener Schichten von Gehirnscans für jeden Patienten durchgeführt werden. In dieser Datenbank können Sie auf die Dateien von 20 verschiedenen Patienten mit Glioblastom vor und nach der Behandlung zugreifen:
3. Der Schwerpunkt dieses Programms und die verschiedenen Methoden, die dieses Projekt leiten, werden in diesem Forschungspapier beschrieben:
Schritt 1: Grafische Benutzeroberfläche (GUI) starten
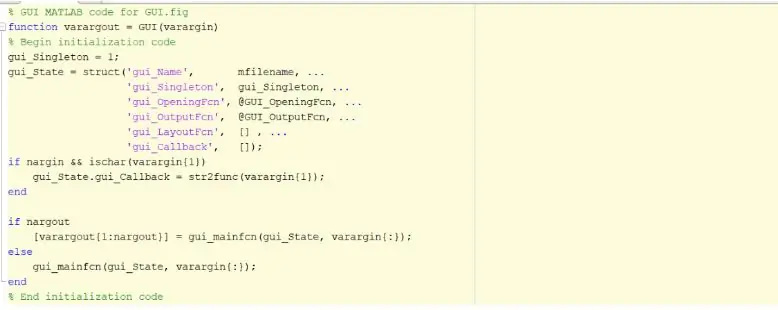
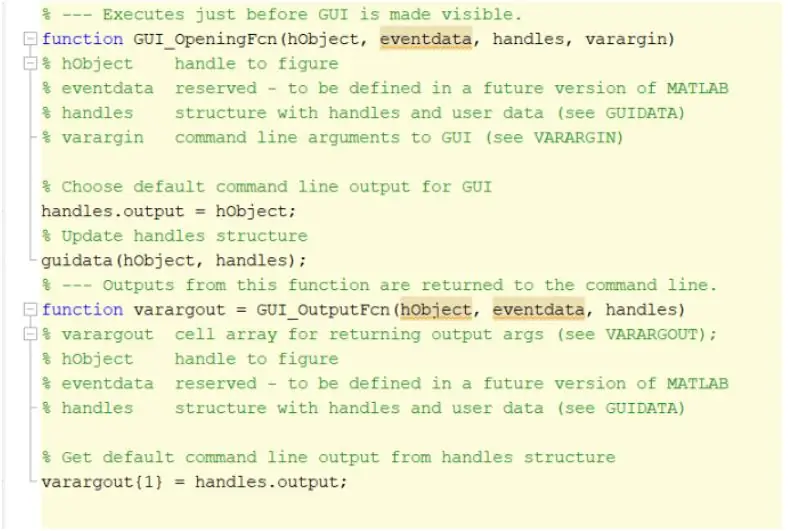
Der erste Schritt besteht darin, die grafische Benutzeroberfläche GUI zu erstellen und zu initiieren. Dies kann durch Eingabe von guide in das Befehlsfenster, Drücken der Eingabetaste und Erstellen einer neuen GUI erfolgen. Sobald dieser Schritt abgeschlossen ist, können Sie mit der Erstellung von Funktionen wie Achsen, statischem Text, Text bearbeiten und Schaltflächen beginnen, die angezeigt werden, sobald das Programm ausgeführt wird und der Benutzer damit interagieren kann. Diese Funktionen können über den Eigenschafteninspektor bearbeitet und manipuliert werden. Das wichtigste Merkmal, das beim Erstellen dieser Funktionen jedoch geändert werden muss, ist der Tag-Name. Es ist wichtig, den Tag-Namen jeder implementierten Funktion zu ändern, da wir dadurch eine unterscheidbare Rückruffunktion erstellen können. Sobald Sie mit dem Layout Ihrer GUI zufrieden sind, können Sie mit dem Laden der DICOM-Dateien fortfahren, die in der GUI angezeigt werden.
Schritt 2: Laden und Lesen von MRT-Bildern in MATLAB
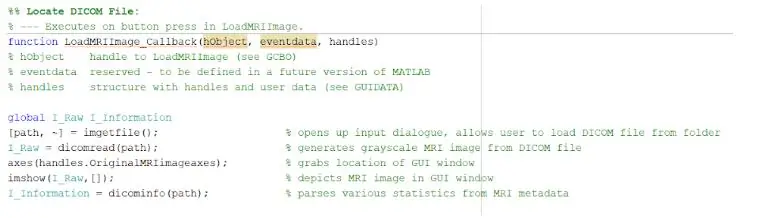
Um die DICOM-Dateien zu laden, müssen Sie die Callback-Funktion, die beim Drücken des Buttons „MRT-Bild laden“ausgeführt wird, korrekt initialisieren. Sobald dies abgeschlossen ist, müssen Sie eine globale Variable erstellen, die das Bild auf den Griffachsen anzeigt, auf denen das Original-MRT-Bild angezeigt werden soll. Die aus der Datenbank heruntergeladenen MRT-Scan-Bilder sind alle Dateien im DICOM-Format, die in Ihr MATLAB-Verzeichnis geladen werden müssen. Suchen Sie die Datei mit imgetfile, um sie in das Programm zu laden. Die Bilder werden mit der eingebauten MATLAB-Funktion ‚dicomread‘gelesen und das erste Rohbild für jede Datei wird mit imshow in die linken GUI-Achsen eingebettet.
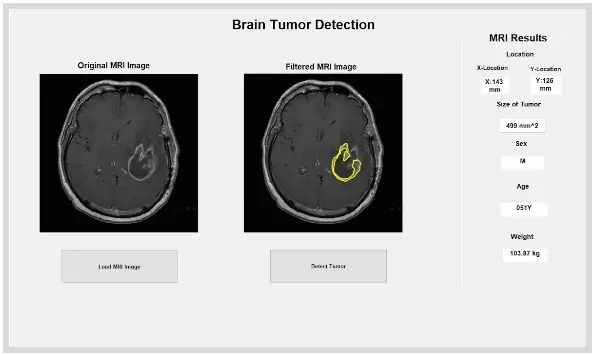
Die eingebaute MATLAB-Funktion „dicominfo“ist auch äußerst nützlich, um alle Informationen jeder MRT-Dicom-Datei zu adressieren. Wir nutzten diese Funktion, um alle beschreibenden Informationen der Patienten wie Geschlecht, Alter, Gewicht und Größe zu extrahieren. Diese Funktion liefert Ihnen auch die Stack-Reihenfolge, die für die Implementierung des Programms innerhalb der grafischen Benutzeroberfläche nützlich ist. Wir haben Variablen für jede der beschreibenden Informationen der Patienten erstellt, die für die GUI verwendet werden, wenn die Erkennungstaste gedrückt wird.
Schritt 3: Bildfilterung

Nachdem die DICOM-Datei des Rohbildes geladen und gelesen wurde, muss das Bild von Graustufen in eine binarisierte Form umgewandelt werden, die nur aus schwarzen und weißen Pixeln besteht Steuern von Aspekten der adaptiven Schwellenwertbildung bei einem Empfindlichkeitswert von 0,59. Der Standardschwellenwert-Empfindlichkeitsfaktor von 0,5 war niedrig und konnte die helleren Blobs und Flecken im Bild nicht erkennen, daher haben wir ihn auf 0,59 erhöht.
Das binarisierte Bild wird dann mit der Funktion „medfilt2“durch einen Medianfilter verarbeitet, da das binarisierte Bild zweidimensional ist. Wir stellen jedes Ausgabepixel so ein, dass es den Medianwert in der 5 x 5 Nachbarschaft um das entsprechende Pixel im binarisierten Eingabebild enthält. Dies reduziert das Rauschen und bewahrt die Kanten in einem 5 x 5 Quadrat um jedes Pixel. Als nächstes wenden wir ein Schiebefenster unter Verwendung von „strel“an, um ein scheibenförmiges flaches Strukturierungselement mit einem Nachbarschaftsradius von 2 zu erzeugen, um jedes zentrale Ursprungspixel in jeder Scheibennachbarschaft zu identifizieren. Wir haben ein Scheibenstrukturierungselement verwendet, da wir jeden kreisförmigen Punkt und die Pixel innerhalb jedes Punkts analysieren, sodass ein scheibenförmiges Element nützlicher ist.
Nachdem das Bild gefiltert wurde, kann es mit der Funktion „imclose“gereinigt werden, um die schwarzen Flecken zwischen den gefilterten weißen Pixeln im Bild zu entfernen und alle Lücken um das Bild zu schließen. Das fertig bearbeitete Bild kann dann im zweiten Subplot der vorab zugewiesenen Figur geplottet werden, was einen Vergleich zwischen dem Rohbild und dem gefilterten Bild ermöglicht.
Schritt 4: Tumorisolierung durch elliptische Maske


Die hellen Flecken des Tumors können dann durch eine vorgenerierte elliptische Maske aus dem gefilterten Hauptbild isoliert werden. Um diese Maske zu erstellen, sollten Sie die Größe des ursprünglichen, rohen MRT-Scan-Bildes kennen und unter Verwendung seiner Zeilen- und Spaltenlänge als x- bzw. y-Koordinaten die Mittelpunktskoordinaten für den Ellipsentrainer zuweisen. Wir legen die y-Achse als Hauptachse mit einem Radius von 50 Einheiten vom Zentrum und die Nebenachse mit einem Radius von 40 Einheiten vom Zentrum fest.
Wir haben die MATLAB-Funktion 'meshgrid' verwendet, um eine kartesische Ebene mit zweidimensionalen Gitterkoordinaten basierend auf den in Vektoren enthaltenen Koordinaten von 1 bis zur Länge der x-Achse und von 1 bis zur Länge der y-Achse des Bildes zu generieren. Col ist eine Matrix, bei der jede Zeile eine Kopie der x-Achse ist, und Row ist eine Matrix, bei der jede Spalte eine Kopie der y-Achse ist. Das kartesische Gitter, das durch die Koordinaten Col und Row repräsentiert wird, hat length(1:Y_Size)-Zeilen und length(1:X_Size)-Spalten. Verwenden Sie die vom kartesischen Gitter erzeugten Indizes von Col und Row, um die Ellipsengleichung in Abhängigkeit vom vorbestimmten Radius und den Mittelpunktskoordinaten zu bestimmen. Der elliptische Umriss kann nun mit den weißen Pixeln gefüllt werden, die von Tumorflecken gefunden wurden.
Unter Verwendung der vorgenerierten elliptischen Maske können wir den spezifischen Tumor, den Sie analysieren möchten, aus dem gefilterten Bild herausschneiden. Die elliptische Maske erkennt, welche Flecken logisch in den Umriss der Ellipse passen und akzeptiert dies als einen Fleck auf dem gefilterten Bild, um als Tumor annehmbar zu sein. Die Funktion „bwareafilt“filtert dann alle anderen Objekte außerhalb dieses erkannten Tumors aus dem Bild heraus. Wir haben empirisch ein spezifisches Fenster von 500 mal 4000 verwendet, basierend auf den Abmessungen aller Bilder. Wir haben dann ein weiteres Schiebefenster mit „Strel“als flaches scheibenförmiges Strukturelement mit einem größeren Nachbarschaftsradius von 6 angewendet, um die Lücken zwischen jedem zentralen weißen Pixel innerhalb des erkannten Tumors zu schließen. Der erkannte Tumorfleck wird mit „imclose“weiter bereinigt, um die schwarzen Pixel weiter zu eliminieren und alle Löcher mit „imfill“zu füllen. Dieser bearbeitete Tumor kann dann im dritten Subplot im vorab zugewiesenen Plot angezeigt werden, um einen Vergleich zwischen dem isolierten Tumor und den ursprünglichen und gefilterten Bildern des MRT-Scans zu ermöglichen.
Schritt 5: Tumorskizze
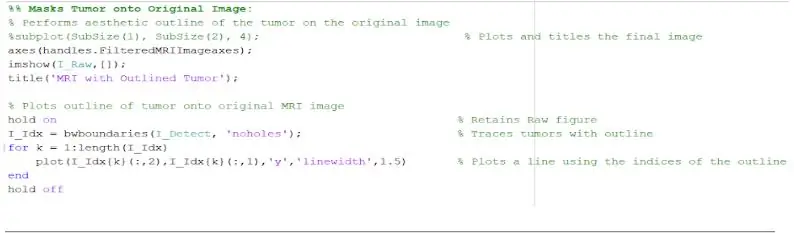
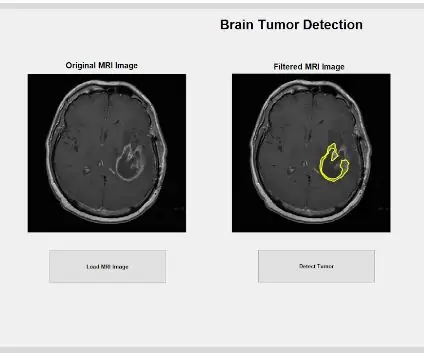
Nachdem der Tumor mit der Maske isoliert wurde, kann er umrissen und auf dem Originalbild angezeigt werden, um seine genaue Lage zu zeigen. Dazu haben wir die Funktion „bwboundaries“verwendet, um den zuvor erkannten Tumor mit einem Umriss nachzuzeichnen. Wir haben den Umriss so festgelegt, dass er die Löcher innerhalb des Tumorobjekts nicht einschließt, während es umrissen wird. Dies kann auf das ursprüngliche Rohbild aufgetragen werden, indem eine „for“-Schleife verwendet wird, die den Umriss um den Tumor mit den Indizes der Linie mit einer Linienbreite von 1,5 Pixel zeichnet. Dieser Umriss wird dann auf das Rohbild aufgetragen und zeigt die genaue Größe und Lage des Tumors relativ zum ursprünglichen MRT-Scan.
Schritt 6: Analyse der physikalischen Eigenschaften von Tumoren
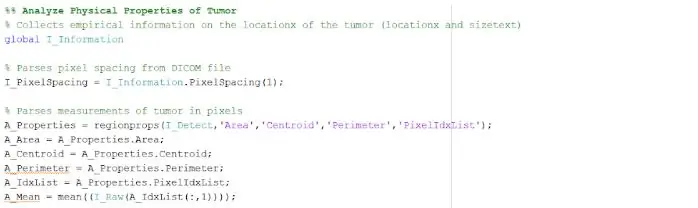
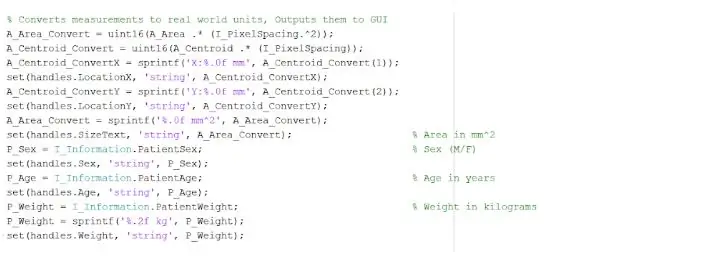
Der isolierte und umrissene Fleck kann uns nützliche Informationen über Größe, Fläche und Lage des Tumors geben. Wir haben die Funktion „regionprops“verwendet, um die Eigenschaften des Tumors zu erkennen, die sich auf Fläche, Umfang, Schwerpunkte und den Pixelindexwert beziehen. Dieser Pixelindexwert gibt uns die realen Einheiten für jedes Pixel jedes Bildes, die für jeden Scan einzigartig sind. Diese Eigenschaften können dann in reale Millimetereinheiten umgerechnet werden. Die empirischen Informationen, die uns das Programm liefert, sind für jede MRT-Untersuchung einzigartig und äußerst nützlich bei der Bestimmung von Größe, Lage und Art des Tumors, die die Benutzer analysieren und in die grafische Benutzeroberfläche integrieren können.
Empfohlen:
Heimwerken -- Einen Spinnenroboter herstellen, der mit einem Smartphone mit Arduino Uno gesteuert werden kann – wikiHow

Heimwerken || Wie man einen Spider-Roboter herstellt, der mit einem Smartphone mit Arduino Uno gesteuert werden kann: Während man einen Spider-Roboter baut, kann man so viele Dinge über Robotik lernen. Wie die Herstellung von Robotern ist sowohl unterhaltsam als auch herausfordernd. In diesem Video zeigen wir Ihnen, wie Sie einen Spider-Roboter bauen, den wir mit unserem Smartphone (Androi
Bewegungsaktivierte Cosplay Wings mit Circuit Playground Express - Teil 1: 7 Schritte (mit Bildern)

Bewegungsaktivierte Cosplay-Flügel mit Circuit Playground Express - Teil 1: Dies ist Teil 1 eines zweiteiligen Projekts, in dem ich Ihnen meinen Prozess zur Herstellung eines Paars automatisierter Feenflügel zeige. Der erste Teil des Projekts ist der Mechanik der Flügel, und der zweite Teil macht es tragbar und fügt die Flügel hinzu
Raspberry Pi mit Node.js mit der Cloud verbinden – wikiHow

So verbinden Sie Raspberry Pi mit Node.js mit der Cloud: Dieses Tutorial ist nützlich für alle, die einen Raspberry Pi mit Node.js mit der Cloud, insbesondere mit der AskSensors IoT-Plattform, verbinden möchten. Sie haben keinen Raspberry Pi? Wenn Sie derzeit keinen Raspberry Pi besitzen, empfehle ich Ihnen, sich einen Raspberry Pi zuzulegen
DIY Circuit Activity Board mit Büroklammern - HERSTELLER - STEM: 3 Schritte (mit Bildern)

DIY Circuit Activity Board mit Büroklammern | HERSTELLER | STEM: Mit diesem Projekt können Sie den Weg des elektrischen Stroms ändern, um durch verschiedene Sensoren zu laufen. Mit diesem Design können Sie zwischen dem Aufleuchten einer blauen LED oder dem Aktivieren eines Summers wechseln. Sie haben auch die Wahl, einen lichtabhängigen Widerstand mit
So erstellen Sie mit MATLAB einen Hörtest für Erwachsene – wikiHow

So erstellen Sie einen Hörtest für Erwachsene mit MATLAB: HAFTUNGSAUSSCHLUSS: Unser Test ist KEINE medizinische Diagnose und sollte nicht als solche verwendet werden. Um das Gehör genau zu messen, wenden Sie sich bitte an einen Arzt. Mit Materialien, die wir bereits hatten, führte unsere Gruppe einen Hörtest durch. Unser Test ist nur für Erwachsene und Jugendliche bestimmt
