Inhaltsverzeichnis:
- Schritt 1: Mammogrammdaten organisieren
- Schritt 2: Bildverarbeitung
- Schritt 3: Schwellenwertcode
- Schritt 4: Auffinden von Anomalien für jedes binäre Bild
- Schritt 5: Plotten der diagnostizierten Massenposition und -größe für den visuellen Vergleich
- Schritt 6: Implementieren der zweiten Vergleichsmethode
- Schritt 7: Analysieren der gesammelten Daten
- Schritt 8: Erstellen Sie Ihren eigenen Klassifikator
- Schritt 9: Verbesserungen? Irgendwelche Gedanken?
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 07:19.
- Zuletzt bearbeitet 2025-01-23 12:52.
Der Zweck dieses Projekts bestand darin, einen Parameter zu identifizieren und zu verwenden, um Graustufen-Mammogrammbilder verschiedener Hintergrundgewebeklassifikationen zu verarbeiten: Fettgewebe, Fettdrüsen und dichtes Gewebe. Diese Klassifizierung wird verwendet, wenn Radiologen Mammographien analysieren und berücksichtigen müssen, ob die Gewebedichte Anomalien wie Läsionen oder Tumoren verdeckt. Dies liegt daran, dass sowohl normale physiologische Strukturen wie Drüsengewebe als auch faseriges Bindegewebe vorhanden sind. und abnorme Morphologien wie Verkalkungen und Tumoren erscheinen auf dem Mammogramm sehr hell, während weniger dichtes Fettgewebe schwarz erscheint. Daher war es angemessen, einen Klassifikator zu programmieren, der Pixelintensitätsniveaus manipulieren kann, um Massen am besten sichtbar zu machen und zu identifizieren.
Schritt 1: Mammogrammdaten organisieren
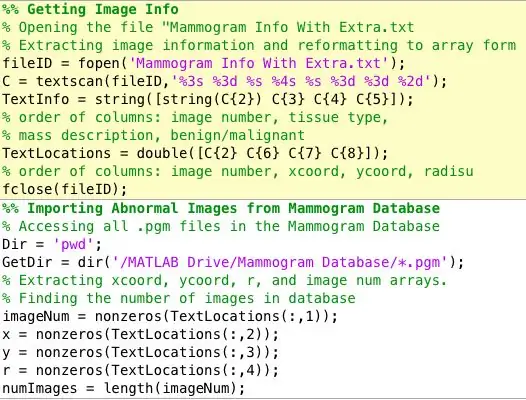
Eines der ersten Dinge, die mir klar wurden, war, die Daten auf eine sehr klare, prägnante und zugängliche Weise zu organisieren. Dies sind die Variablen, die ich aus der Mini-MIAS-Datenbank für Mammogramme extrahiert habe. Ich habe zwei Arrays erstellt. Eine mit 4 Spalten:
- Bildnummer:
- x-Koordinate der Masse
- y-Koordinate der Masse
- Massenradius: (Dies definiert eine ungefähre Größe für die Masse
Das zweite Array enthielt Klassifizierungsinformationen:
- Art des Hintergrundgewebes: Fett (F), Fettdrüse (G), Dicht (D)
- Beschreibung der Masse: Wohldefiniert (CIRC), spiculated (SPIC), schlecht definiert andere (MISC) Architekturverzerrung (ARCH), Asymmetrie (ASYM), Normal (NORM)
- Diagnosen: Gutartig (B), Bösartig (M)
Da das Ziel dieses Projekts darin bestand, den besten Schwellenwert für jede Art von Hintergrundgewebe zu bestimmen, waren nicht alle Informationen erforderlich. Sie können Ihr Projekt jedoch um Texturanalysen erweitern und Ihren Klassifikator gegen die bekannten Massenbeschreibungen testen.
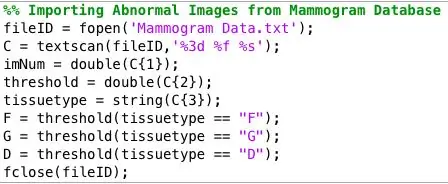
Randnotiz: Die Datenbank, aus der ich die diagnostizierten Mammogrammbilder erhalten habe, organisierte die Informationen zu jedem Mammogramm in einer von den Bildern getrennten Textdatei. Es war etwas schwierig für mich, die Daten aus einer Textdatei zu extrahieren und in Array-Formularen zu organisieren, aber der folgende Link war sehr hilfreich, um all das herauszufinden. Alternativ passen Sie einfach den Code, den ich oben eingefügt habe, für Ihre Zwecke an.
Mammogramm-Dateiformat: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
TextScan-Hilfe:
Schritt 2: Bildverarbeitung
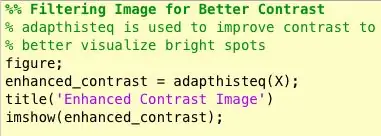
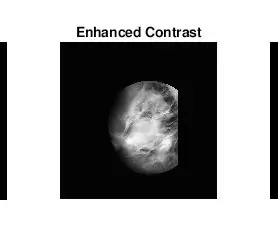
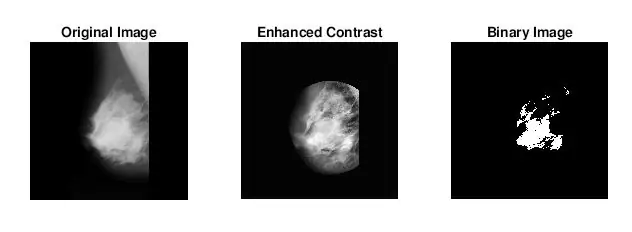
Nun, das zweite, was mir auffiel, als ich herausfand, wie man Massen identifiziert, war, dass ich bei vielen anormalen Mammogrammen nicht visuell erkennen konnte, wo die Anomalie war oder wie groß sie war. Da ich kein erfahrener Radiologe bin, wurde es natürlich erwartet. Der einfachste Weg, Anomalien zu finden (laut meiner langen Google-Suche), bestand jedoch darin, Konzentrationen von hellen und dunklen Bereichen zu untersuchen. Ich habe hauptsächlich die Funktion adapthisteq verwendet, um den Bildkontrast zu verbessern, und dann imbinarisieren, um das Bild in ein Binärbild umzuwandeln, um mit verschiedenen Schwellenwerten zu experimentieren.
- adapthisteq: Diese Funktion transformiert Intensitätswerte von Graustufen- und RGB-Bildern mit kontrastbegrenzter adaptiver Histogramm-Entzerrung. Mit anderen Worten, es passt das Histogramm der Intensitätswerte an einen bestimmten Verteilungstyp an. Der Mathworks-Link für diese Funktion ist unten zum Weiterlesen angehängt.
- imbinarize: Erstellt ein Binärbild aus einem Graustufenbild, indem alle Pixel über einer bestimmten Intensität 1s und die Pixel unter diesem Wert eine 0 zugewiesen werden. Ich habe diese Funktion verwendet, um den optimalen Schwellenwert zur Reduzierung des Hintergrundgeweberauschens zu testen.
Schritt 3: Schwellenwertcode
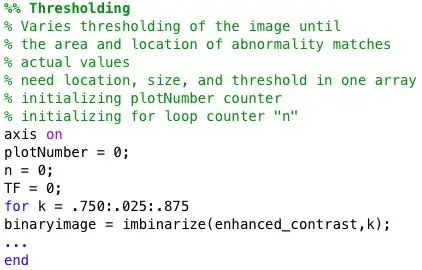
Eine for-Schleife wird verwendet, um das Mammogramm mit unterschiedlichen Schwellenwerten zu binarisieren. Um ein größeres Bild zu erhalten, enthält die for-Schleife den Code von Schritt 3 bis Schritt 7. Jedes Binärbild wird also auf Auffälligkeiten analysiert. Darüber hinaus ist diese for-Schleife in eine andere for-Schleife eingeschlossen, die in jeder Iteration ein neues Mammogramm-Bild aus der Datenbank importiert.
Schritt 4: Auffinden von Anomalien für jedes binäre Bild
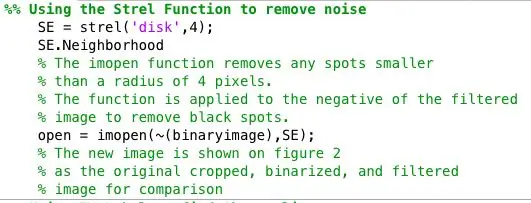
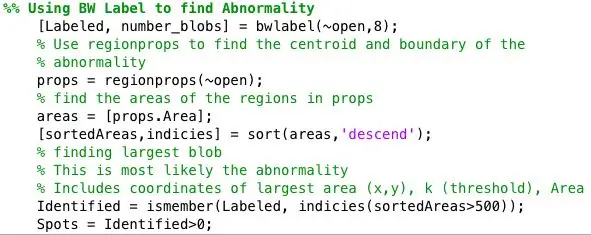
Ich habe die Binärbilder mit der Strel-Funktion in Verbindung mit imopen weiterverarbeitet, um Hintergrundrauschen zu entfernen. Das Binärbild aus dem vorherigen Schritt wird invertiert und unter Verwendung der durch SE definierten Nachbarschaft gefiltert. Dann habe ich bwlabel verwendet, um alle Bereiche zu beschriften, die mindestens 8 verbundene Pixel hatten.
Die Funktion "Region Props" wurde verwendet, um die Schwerpunkt- und Flächeneigenschaften jedes von bwlabel identifizierten Spots zu finden.
Dann wurden alle Spots größer als 500 Pixel mit ismember identifiziert. Die Schwerpunkte für die identifizierten Spots wurden auf einem Bild aufgetragen, das nur die Spots mit einer größeren Fläche als 500 anzeigte. Flecken = Identifiziert>0;
Schritt 5: Plotten der diagnostizierten Massenposition und -größe für den visuellen Vergleich
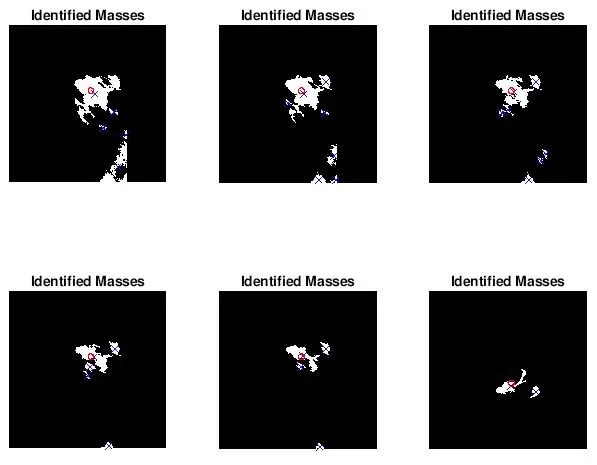
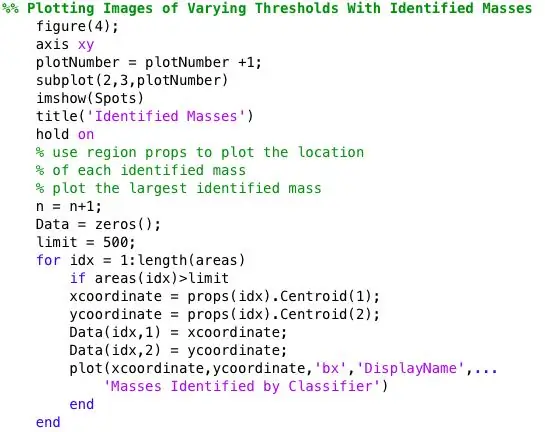

Ich wollte sehen, ob die von bwlabel gefundenen Spots richtig sind. Ich habe dies auf zwei Arten getan. Ich habe zuerst die Genauigkeit meines Klassifikators analysiert, indem ich einen visuellen Vergleich durchgeführt habe. Ich habe einfach die tatsächliche Größe und Position der Anomalie (roter Kreis) und die durch den Code (blaues x) bestimmte Position auf dem vorverarbeiteten Mammogramm-Bild aufgetragen. Die sechs obigen Bilder zeigen die Auswirkungen einer Erhöhung des Graustufenschwellenwerts.
Schritt 6: Implementieren der zweiten Vergleichsmethode

Die zweite Möglichkeit, den Klassifikator und die Schwellenwerte zu testen, bestand darin, zu bestimmen, ob die vom Klassifikator gefundenen Orte innerhalb einer bestimmten Entfernung von den Koordinaten der diagnostizierten Anomalie lagen. Ich habe die Schwellenwerte, bei denen mindestens einer der identifizierten Punkte innerhalb von 1,5 * r von der bekannten Anomalie lag, in einer separaten Textdatei namens Mammogrammdaten gespeichert. Der Zweck dafür war, den minimalen Schwellenwert zu finden, der für meinen Klassifikator erforderlich ist, um die Anomalie zu identifizieren.
Schritt 7: Analysieren der gesammelten Daten
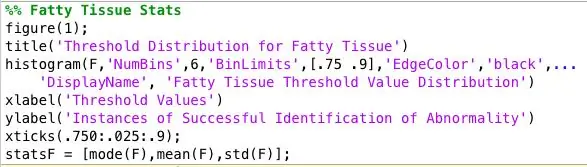
Ich habe das Programm auf allen abnormalen Mammogrammbildern ausgeführt und ich hatte eine riesige Textdatei mit Daten. Um den besten Schwellenwert für jeden Gewebetyp zu finden, ordnete ich die Daten nach Gewebetyp und zeichnete ein Histogramm der Schwellenwerte für jeden Gewebetyp. Der richtige Schwellenwert wurde entschieden, welcher Schwellenwert die genauesten Ergebnisse für jeden Gewebetyp lieferte. Ich habe diese Daten gespeichert, um sie in meinen Klassifikator hochzuladen.
Schritt 8: Erstellen Sie Ihren eigenen Klassifikator

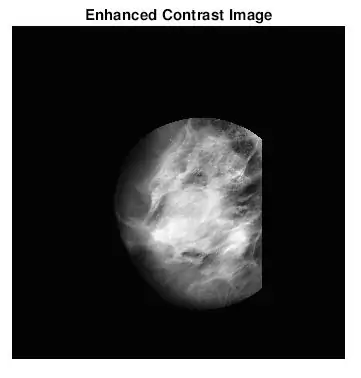
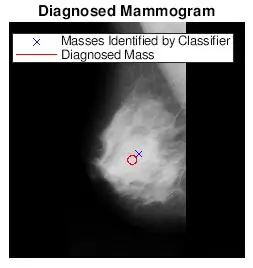
Nachdem ich die am besten geeigneten Schwellenwerte für jeden Gewebetyp gefunden hatte, bearbeitete ich meinen ursprünglichen Code, damit ein Benutzer die Bildnummer und den Gewebetyp eingab, um den Schwellenwert für das Mammogrammbild auszuwählen. Ich habe dann die diagnostizierte Mammogramm-Position mit den gefundenen Positionen auf den Original-Mammogramm-Bildern aufgetragen. Ich wollte dies mehr Spaß machen, also habe ich eine Funktion programmiert, um einen kreisförmigen Bereich um den ROI herum zuzuschneiden. Der Benutzer wird angewiesen, einen Mittelpunkt und mehrere Punkte auszuwählen, die den ROI am besten umfassen. Ich habe beide Matlab-Dateien hier angehängt.
Schritt 9: Verbesserungen? Irgendwelche Gedanken?
Als ich dieses anweisbare schrieb, beginne ich, viele Verbesserungen zu sehen, die ich am Klassifikator vornehmen könnte, z. Datei. Da dies ein Projekt mit einer Deadline war, musste ich irgendwo aufhören, aber ich hoffe, dass ich die Bildverarbeitungskenntnisse, die ich in anderen Anwendungen erlernt habe, anwenden kann. Außerdem habe ich die Datei angehängt, die verwendet wurde, um alle anormalen Mammogrammbilder stapelweise zu verarbeiten.
Empfohlen:
So richten Sie einen Raspberry Pi ein und verwenden ihn – wikiHow

So richten Sie einen Raspberry Pi ein und verwenden ihn: Für die Leser der Zukunft sind wir im Jahr 2020. Das Jahr, in dem Sie, wenn Sie das Glück haben, gesund zu sein und nicht mit Covid-19 infiziert sind, plötzlich , hat viel mehr Freizeit, als du dir jemals vorgestellt hast. Wie kann ich mich also nicht zu dumm beschäftigen? Oh ja
So verwenden Sie Fritzing, um eine Leiterplatte zu erstellen – wikiHow
So verwenden Sie Fritzing, um eine Leiterplatte herzustellen: In diesem Instructable zeige ich Ihnen, wie Sie Fritzing verwenden. In diesem Beispiel werde ich einen Energieschild für Arduino erstellen, mit dem das Arduino mit einer Batterie mit Strom versorgt werden kann
So verwenden Sie einen PIR-Sensor und ein Summermodul - Visuino-Tutorial: 6 Schritte

So verwenden Sie einen PIR-Sensor und ein Summermodul - Visuino-Tutorial: In diesem Tutorial lernen wir, wie Sie einen PIR-Sensor und ein Summermodul verwenden, um jedes Mal, wenn ein PIR-Sensor eine Bewegung erkennt, ein Geräusch zu erzeugen. Sehen Sie sich ein Demonstrationsvideo an
Flussmittel verwenden - Lötgrundlagen: 5 Schritte

Flussmittel verwenden | Grundlagen des Lötens: Wenn Sie löten, muss das Lot eine gute Verbindung zu den Teilen herstellen, auf die Sie löten. Das Metall der Teile und das Metall des Lotes müssen direkt miteinander in Kontakt kommen, um eine gute Verbindung herzustellen. Aber da ich
So verwenden Sie den alten LCD-Bildschirm Ihres defekten Laptops wieder – wikiHow

So verwenden Sie den alten LCD-Bildschirm Ihres kaputten Laptops: Dies ist ein super einfaches, aber auch sehr tolles Projekt. Mit der richtigen Treiberplatine können Sie jeden modernen Laptop-Bildschirm in einen Monitor verwandeln. Die Verbindung dieser beiden ist ebenfalls einfach. Einfach Kabel einstecken und fertig. Aber ich ging noch einen Schritt weiter und auch b
