Inhaltsverzeichnis:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 07:15.
- Zuletzt bearbeitet 2025-01-23 12:52.
Von: Phuc Lam, Paul Yeung, Eric Reyes
Die Erkenntnis, dass Fehler bei der Segmentierung der Lunge zu falschen Informationen bezüglich der Identifizierung eines Krankheitsbereichs führen und sich direkt auf den Diagnoseprozess auswirken können. Moderne computergestützte Techniken lieferten keine genauen Ergebnisse, wenn Lungenerkrankungen schwierige Formen haben. Diese abnormalen Formen können durch Pleuraergüsse, Konsolidierungen usw. verursacht werden. Durch die Anwendung der Lungensegmentierungstechnik, bei der die Grenzen der Lunge vom umgebenden Brustgewebe isoliert werden, kann unsere App die Grenzen mit den Eingabeschwellen des Benutzers identifizieren, um vollständig anpassbare Ansichten zu erhalten der Formen der Lunge, Der Zweck dieses MatLab-Projekts besteht darin, eine benutzerfreundliche interaktive Lungensegmentierungs-App zu erstellen, um pathologische Zustände der Röntgenbilder der Lunge zu erkennen. Unser Ziel ist es, eine effektivere Möglichkeit zu schaffen, abnormale Lungen darzustellen und zu identifizieren, um Ärzten und Radiologen eine zuverlässigere Möglichkeit zu geben, Lungenerkrankungen zu diagnostizieren. Mit dem App-Designer-Tool in MatLab wurde das Programm speziell für die Arbeit mit Thoraxröntgen und Computertomographie (CT) entwickelt, aber es wurde auch für MRT-Scans getestet.
Die folgende Anleitung enthält unsere Rauschfiltertechnik (Tiefpass-Wiener-Filter) sowie die Bildschwelle (unter Verwendung des Intensitätshistogramms des Graustufenbildes) und die Verwendung eines morphologischen Gradienten (der Unterschied zwischen der Dilatation und der Erosion eines Bildes) um eine Region von Interesse identifizieren. In der Anleitung wird dann erklärt, wie wir alle Elemente in die grafische Benutzeroberfläche (GUI) integrieren.
Notiz:
1). Dieses Projekt ist inspiriert von einem Forschungspapier: "Segmentation and Image Analysis of Abnormal Lungs at CT: Current Approaches, Challenges, and Future Trends". Welche finden Sie hier
2). Wir verwenden Röntgenbilder von NIH: Clinical Center. Link findet ihr hier
3). Hilfe zum App-Designer finden Sie hier
4). Bevor Sie den Code ausführen: Sie müssen den Dir-Pfad (in Zeile 34) auf Ihr Dateiverzeichnis und den Bildtyp (Zeile 35) ändern (wir analysieren *.png).
Schritt 1: Schritt 1: Bild laden
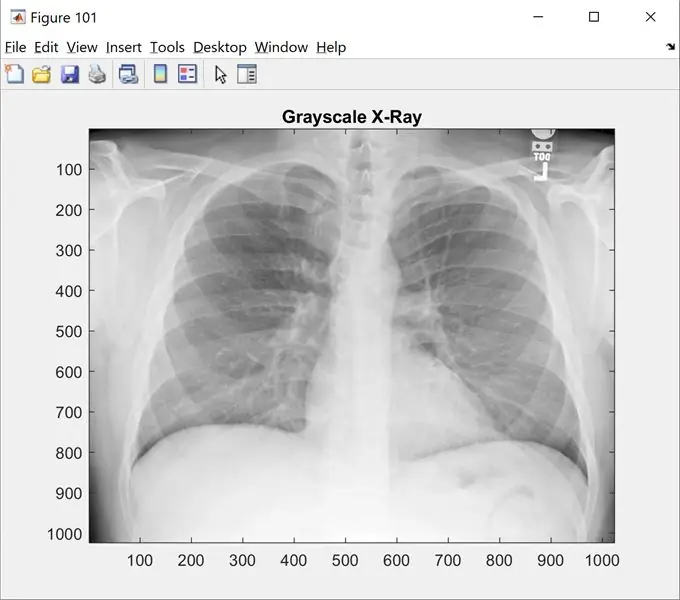
Dieser Schritt zeigt Ihnen das Originalbild in Graustufen. Ändern Sie 'name_of_picture.png' in Ihren Bildnamen
klar; clc; alle schließen;
%% Bilder werden geladen
raw_x_ray='name_of_picture.png';
I=imread(raw_x_ray);
Abbildung (101);
imshow(I);
Farbkarte (grau);
title('Graustufen-Röntgenbild');
Schritt 2: Schritt 2: Rauschfilterung und Histogramm
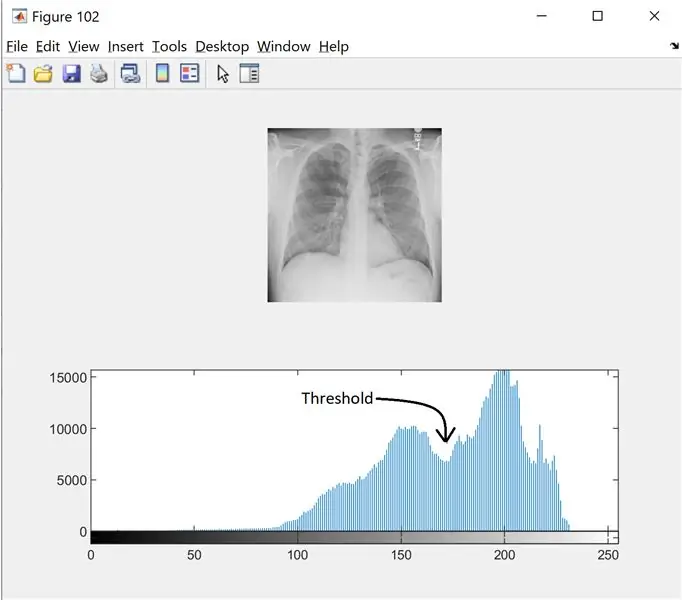
Um den Schwellenwert für das Graustufenbild zu finden, schauen wir uns das Histogramm an, um zu sehen, ob es sich um unterschiedliche Modi handelt. Lesen Sie hier mehr
I=wiener2(I, [5 5]);
Abbildung (102);
Nebenhandlung (2, 1, 1);
imshow(I);
Nebenhandlung (2, 1, 2);
imhist(I, 256);
Schritt 3: Schritt 3: Schwellenwerte festlegen
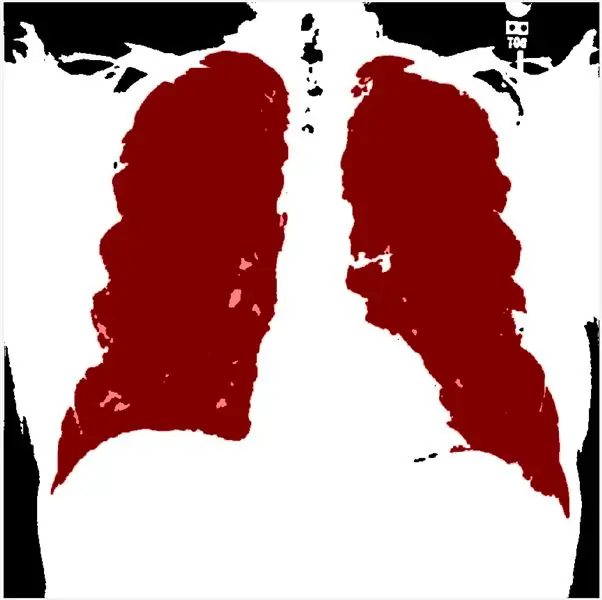
In diesem Schritt können Sie den Schwellenwert gemäß dem Histogramm einstellen. morphologicalGradient hebt den interessierenden Bereich rot hervor, und die Funktion visboundaries überlagert das umrissene und gefilterte Bild der Lunge rot.
Durch die Verwendung von Regionprops können wir die Solidility-Arrays bestimmen und sie absteigend sortieren. Als nächstes binarisiere ich das Graustufenbild und wende die morphologische Gradientenmethode und mloren Shurasking an, um die Region of Interest (ROI) hervorzuheben. Der nächste Schritt besteht darin, das Bild umzukehren, sodass der Lungen-ROI weiß auf schwarzem Hintergrund ist. Ich verwende die Funktion showMaskAsOverlay, um 2 Masken anzuzeigen. Hinweis: Der Code ist inspiriert von Loren Shure, Link.
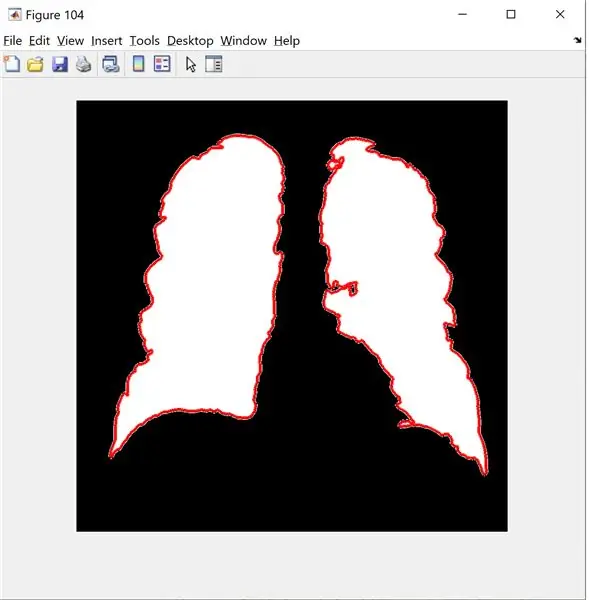
Schließlich erzeuge ich einen roten Umriss, indem ich die bwbwboundaries verwende und das Filterbild und die Grenzen maskiere.
a_thresh = ich >= 172; % setzen Sie diesen Schwellenwert
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
props = regionprops(a_thresh, 'alle');
sortedSolidity = sort([props. Solidity], 'absteigen');
SB = sortierte Solidität(1);
wenn SB == 1 % SB nur Festigkeit akzeptieren == 1 Knochen herausfiltern
binaryImage = imbinarize(I); Abbildung (103);
imshow(binaryImage); Farbkarte (grau);
SE = strel('Quadrat', 3);
morphologicalGradient = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
mask = imbinarisieren (morphologischer Gradient, 0.03);
SE = strel('Quadrat', 2);
Maske = imclose(Maske, SE);
mask = imfill(maske, 'Löcher');
Maske = bwareafilt (Maske, 2); % Kontrollnummer der Bereichsshow
notMask = ~maske;
Maske = Maske | bwpropfilt(notMask, 'Bereich', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, Maske, 'r'); % Sie müssen die App/Funktion showMaskAsOverlay herunterladen
BW2 = imfill(binaryImage, 'Löcher');
neues_image = BW2;
neues_bild(~maske) = 0; % invertieren Hintergrund und Löcher
B=bwboundaries(new_image); % kann nur 2 Dimensionen akzeptieren
Abbildung (104);
imshow(neues_bild);
festhalten
grenzüberschreitende (B);
Ende
Schritt 4: GUI erstellen
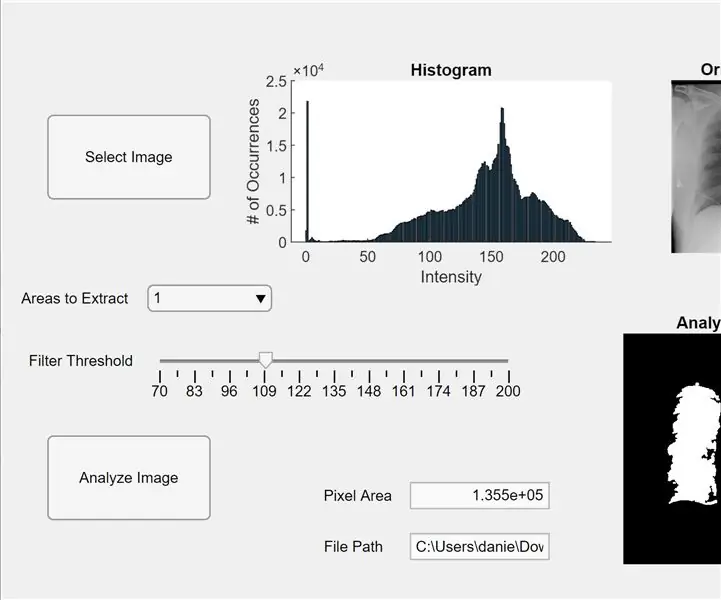
Jetzt integrieren wir den vorherigen Code in eine MATLAB-App. Öffnen Sie den App Designer in MATLAB (Neu > App). Zuerst gestalten wir die Benutzeroberfläche durch Klicken-Halten-und Ziehen in drei Achsen in den mittleren Arbeitsbereich. Als nächstes klicken und halten wir zwei Schaltflächen, ein Bearbeitungsfeld (Text), ein Bearbeitungsfeld (numerisch), einen Schieberegler und ein Dropdown-Menü. Zwei Achsen zeigen jeweils die Vorschau an und analysiert das Bild, und die dritte Achse zeigt ein Histogramm von Pixeln für das "ausgewählte" Vorschaubild an. Das Bearbeitungsfeld (Text) zeigt den Dateipfad des ausgewählten Bildes an und das Bearbeitungsfeld (numerisch) zeigt den erkannten Pixelbereich der Lunge an.
Wechseln Sie nun im App Designer von der Entwurfsansicht in die Codeansicht. Geben Sie im Code den Code für die Eigenschaften ein, indem Sie auf die rote Schaltfläche "Eigenschaften" mit einem Pluszeichen klicken. Initialisieren Sie die Eigenschaften I, Threshold und RegionsToExtract wie im unten angegebenen Code. Klicken Sie als Nächstes mit der rechten Maustaste auf eine Schaltfläche oben rechts im Arbeitsbereich (dem Komponentenbrowser) und gehen Sie zu Rückrufe>Gehe zu… Rückruf. Fügen Sie den Code für „Funktion SelectImageButtonPushed(app, event)“hinzu. Mit diesem Code können Sie ein Bild auswählen, das von Ihrem Computer mit uigetfile analysiert werden soll. Nach der Auswahl eines Bildes erscheint unter den Achsen ein Vorschaubild zusammen mit einem Histogramm. Klicken Sie dann mit der rechten Maustaste auf die andere Schaltfläche und wiederholen Sie das gleiche Verfahren, um eine Rückruffunktion zu erstellen.
Fügen Sie den Code unter "function AnalyzeImageButtonPushed(app, event)" hinzu. Dieser Code führt die Pixelzählung und Blob-Erkennung für das Vorschaubild bei der Schaltfläche "Bild analysieren" durch (unabhängig davon, welche Schaltfläche Sie für diesen Code mit der rechten Maustaste angeklickt haben). Nach der Programmierung der Tasten programmieren wir nun den Schieberegler und das Dropdown-Menü. Klicken Sie mit der rechten Maustaste auf den Schieberegler, erstellen Sie eine Callback-Funktion und fügen Sie den Code unter „function FilterThresholdSliderValueChanged(app, event)“bis zum Ende ein. Dadurch kann der Schieberegler den Schwellenwert für die Grauintensität anpassen.
Erstellen Sie eine Rückruffunktion für das Dropdown-Menü und fügen Sie den Code unter „Funktion AreastoExtractDropDownValueChanged(app, event)“hinzu, damit das Dropdown-Menü die Anzahl der auf den analysierten Bildachsen angezeigten Blobs ändern kann. Klicken Sie nun auf jedes Element im Komponentenbrowser und ändern Sie deren Eigenschaften nach Ihren Wünschen, z. B. Ändern der Namen der Elemente, Entfernen von Achsen und Ändern der Skalierung. Ziehen Sie die Entitäten des Komponentenbrowsers in der Entwurfsansicht per Drag & Drop in ein funktionales und leicht verständliches Layout. Sie haben jetzt eine App in MATLAB, die Bilder von Lungen für den Pixelbereich analysieren kann!
Eigenschaften (Zugriff = privat)I = ; % Bilddatei
Schwelle = 257; %Schwellenwert für die Binarisierung der Grauintensität
regionToExtract = 2;
Ende
Funktion SelectImageButtonPushed(App, Ereignis)
clc;Dir = 'C:\Benutzer\danie\Downloads\images_004\images'; %define invariate Datei "Präfix"
[imageExt, Pfad] = uigetfile('*.png'); %nehmen Sie den variablen Teil des Bildnamens
imageName = [Dir filesep imageExt]; % verketten invariate und variable Belastungen
app. I = imread(imageName); %das Bild lesen
imshow(app. I, 'Eltern', app. UIAxes); %das Bild anzeigen
app. FilePathEditField. Value = Pfad; % Dateipfad anzeigen, woher das Originalbild stammt
Ende
Funktion AnalyzeImageButtonPushed(App, Ereignis)
originalImage = app. I;
originalImage = wiener2(app. I, [5 5]); %Punktentfernungsfilter
Histogramm (app. AchsenHistogramm, app. I, 256); % Histogramm des Bildes anzeigen
a_thresh = originalImage >= app.threshold; % setzen Sie diesen Schwellenwert
labelImage = bwlabel(a_thresh);
props = regionprops(a_thresh, 'alle');
sortedSolidity = sort([props. Solidity], 'absteigen');
SB = sortierte Solidität(1);
wenn SB == 1 % SB nur Festigkeit akzeptieren ==1 Knochen herausfiltern
SE = strel('Quadrat', 3);
morphologicalGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
mask = imbinarisieren (morphologischer Gradient, 0.03);
SE = strel('Quadrat', 2);
Maske = imclose(Maske, SE);
mask = imfill(maske, 'Löcher');
mask = bwareafilt(maske, app.regionsToExtract);
% Kontrollnummer der Bereichsshow
notMask = ~maske;
Maske = Maske | bwpropfilt(notMask, 'Bereich', [-Inf, 5000 - eps(5000)]);
BW2 = imfill(labelImage, 'Löcher');
neues_image = BW2;
neues_bild(~maske) = 0;
B = bwboundaries(new_image); % kann nur 2 Dimensionen akzeptieren imshow(new_image, 'parent', app. UIAxes2);
hold(app. UIAxes2, 'an');
grenzüberschreitende (B);
set(gca, 'YDir', 'umgekehrt');
lungArea = bwarea(new_image);
app. PixelAreaEditField. Value = lungArea;
Ende
Ende
Funktion FilterThresholdSliderValueChanged(App, Ereignis)
app.threshold = app. FilterThresholdSlider. Value;
Ende
Funktion AreastoExtractDropDownValueChanged(app, event)stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
Ende
Ende
Empfohlen:
So programmieren und veröffentlichen Sie Matlab 2016b in Word (Anleitung für Anfänger): 4 Schritte

So codieren und veröffentlichen Sie Matlab 2016b in Word (Einsteigerhandbuch): Matlab ist ein leistungsstarkes Sprachprogramm, das zum Berechnen technischer Ergebnisse verwendet wird. Es hat die Fähigkeit, Visualisierungen, Berechnungen und Programmierung auf benutzerfreundliche Weise zu integrieren. Mit diesem Programm kann der Benutzer Probleme und Lösungen veröffentlichen
Programmiererweiterungen in MATLAB trainieren: 6 Schritte

Verbesserungen bei der Zugprogrammierung in MATLAB: Ziel: Das Ziel dieses programmierten Systems besteht darin, einen kleinen Arduino zu betrachten und die Codierung in größerem Maßstab anzuwenden, um möglicherweise bestimmte Sicherheitsfunktionen von Amtrak-Eisenbahnsystemen zu verbessern. Dazu haben wir einen Bodenfeuchtesensor hinzugefügt
Tutorial zum Abtasten analoger Signale - MATLAB: 8 Schritte

Tutorial zum Abtasten analoger Signale | MATLAB: In diesem Tutorial zeigen wir, was Sampling ist. und Wie man ein analoges Signal mit der MATLAB-Software abtastet
So erstellen Sie mit MATLAB einen Hörtest für Erwachsene – wikiHow

So erstellen Sie einen Hörtest für Erwachsene mit MATLAB: HAFTUNGSAUSSCHLUSS: Unser Test ist KEINE medizinische Diagnose und sollte nicht als solche verwendet werden. Um das Gehör genau zu messen, wenden Sie sich bitte an einen Arzt. Mit Materialien, die wir bereits hatten, führte unsere Gruppe einen Hörtest durch. Unser Test ist nur für Erwachsene und Jugendliche bestimmt
Matlab-Grundlagen: 6 Schritte

Matlab-Grundlagen: Dieses instructable deckt einige der grundlegendsten Funktionen von Matlab ab. Sie werden lernen, wie Sie Matlab dazu bringen, eine periodische Funktion intern auszuführen und zu plotten und wie Sie stattdessen dieselbe periodische Funktion aus einer Excel-Datei ziehen und plotten. Diese Funktionen
